DataFrameの項目間の相関係数を2次元配列形式で表示させる方法です(相関行列)。DataFrameのcorrメソッドを用いればワンライナーで相関行列を表示させることができます。
開発環境
- matplotlib 3.4.2
- pandas 1.3.3
- Python 3.7.11
- seaborn 0.11.2

相関係数とは
相関係数とは2つの項目の関係性の強さを-1~0~+1の範囲で表しまたものです。-1もしくは+1に近づくほど2つの項目には強い関係(強い相関)があると言え、0に近づくほど「関係なし」ということになります。
相関関係の計算方法には以下のようなものがあります。
- Pearsonの積率相関係数
- Kendallの順位相関係数
- Spearmanの順位相関係数
一般的に単に「相関係数」という場合はPearsonの積率相関係数のことを指します。XとYの相関係数(Pearsonの積率相関係数)ρは以下のようになります。
$$ρ=\frac{cov(X,Y)}{σ_X σ_Y} (cov(X,Y):共分散、σ_X, σ_Y:標準偏差)$$
相関係数ρの値の解釈は明確には定まっていませんが、次のような基準が目安となります。
| 0.7 < |ρ| | 強い相関あり |
| 0.4 < |ρ| < 0.7 | 相関あり |
| 0.2 < |ρ| < 0.4 | 弱い相関あり |
| |ρ| < 0.2 | 相関なし |
Pearsonの積率相関係数は分布に正規分布を仮定しているパラメトリックな指標ですが、ノンパラメトリックな指標としてはKendallの順位相関係数やSpearmanの順位相関係数などがあります。
DataFrameの相関行列を計算する方法
DataFrameのcorrメソッドを用いてそのDataFrameの項目間の相関係数を算出して2次元配列形式で表示させることができます。
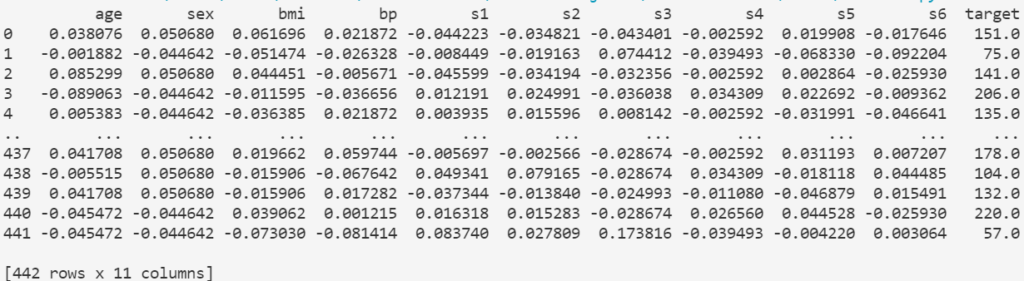
ここでは例としてscikit-learnの「糖尿病の診療についてのデータセット」の相関行列を表示してみましょう。
このデータセットに含まれる項目は以下のようになります。
- age : 年齢
- sex : 性別
- bmi : BMI
- bp : 平均血圧
- s1 : 総コレステロール
- s2 : LDL
- s3 : HDL
- s4 : 総コレステロール / HDL
- s5 : トリグリセリド(対数)
- s6 : GLU(血糖値レベル)
- target : ベースラインから1年後の糖尿病の進行度
そのDataFrameのcorrメソッドを実行することで、相関行列をDataFrameとして取得することができます。
from sklearn import datasets
df = datasets.load_diabetes(as_frame=True).frame
corr = df.corr()
print(corr)
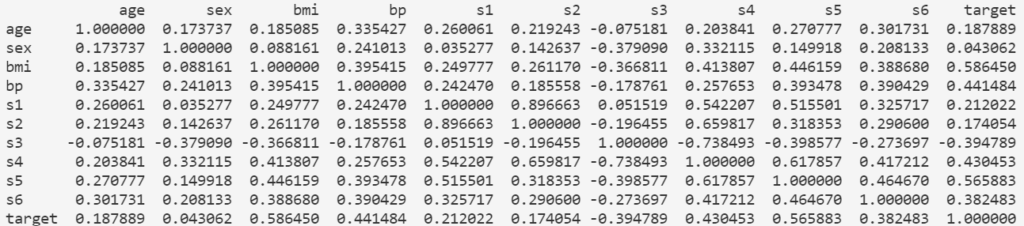
なお、デフォルトで求められる相関係数はPearsonの積率相関係数ですが、以下のように引数に指定することで、Kendallの順位相関係数やSpearmanの順位相関係数を求めることも可能です。
# Kendallの順位相関係数
corr = df.corr('kendall')
# Spearmanの順位相関係数
corr = df.corr('spearman')
相関行列の可視化
ヒートマップの作成
相関行列の可視化にはヒートマップを用いることが一般的です。
それでは、先ほど取得した相関行列を表すcorrをヒートマップで可視化してみましょう。
import seaborn as sns
import matplotlib.pyplot as plt
sns.heatmap(corr, cmap='coolwarm', vmin=-1, vmax=1, annot=True)
plt.show()
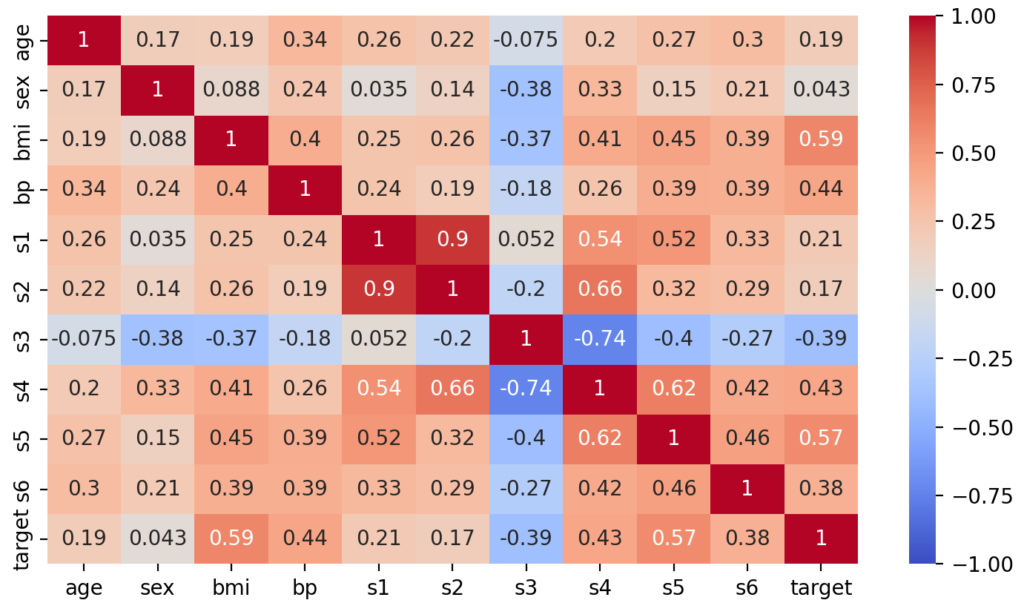
seabornのheatmap関数でヒートマップのカラーバーの範囲は-1~+1に指定して、相関係数の値をセルに書き込んで表示するようにしています。
ヒートマップの読み方
ヒートマップでは行と列の交点のセルにその相関係数が格納されて、右側のカラーバーに従って色分けされています。左上から右下の対角線は同じもの同士の相関係数になるのでその値は1であり、ヒートマップはこの対角線に対して線対称となります。
では、先ほど作成したヒートマップから、「糖尿病の進行度と検査項目との相関関係」だけを抜き出して考えてみましょう(目的変数と説明変数の相関関係)。1番下の行だけを抽出します。
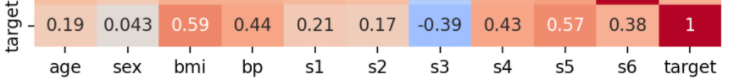
これを見ると、bmiやbp、s4、s5などには正の相関がありそうであることが分かります。また、これらの説明変数の中でs3だけ負の相関となっていることも分かります。










コメント