生存時間解析の基本はカプラン・マイヤー曲線による生存確率関数の可視化です。ここではPythonでカプラン・マイヤー推定量を求めて、それをグラフ化する方法をお示しします。なお、生存時間解析という名前ですが、あるイベントが起こるまでの時間分布についての解析なので、その対象は生存/死亡に限らず、「故障が起こるまでの時間」など様々な分野に使われる分析になります。
開発環境
- lifelines 0.26.3
- matplotlib 3.4.2
- Python 3.7.11

カプラン・マイヤー推定量とは?
観察対象のイベント発生時刻を小さい順に並べて、t(1)、t(2)、…、t(r)と置くとき、カプラン・マイヤー推定量とは、時刻t(i)≦t<t(i+1)の時までにまだイベントが起きていない確率を表します。つまり、生存/死亡について考える場合は、カプラン・マイヤー推定量は寿命がtより長くなる確率(生存確率関数S(t))を表します。
なお、Pythonでカプラン・マイヤー統計量を用いる場合はlifelinesモジュールを用いますが、これはAnacondaではインストールされないので、自分でインストールする必要があります。以下のコマンドでインストールしましょう。
conda install -c conda-forge lifelines
サンプルデータ
ここではlifelinesモジュールのload_larynx関数を用いて、サンプルデータとして含まれている喉頭癌のステージ別・年齢別の生存期間を表すデータを取得します。
from lifelines import datasets
larynx_data = datasets.load_larynx()
print(larynx_data)
time age death Stage_II Stage_III Stage_IV 0 0.6 77 1 0 0 0 1 1.3 53 1 0 0 0 2 2.4 45 1 0 0 0 3 2.5 57 0 0 0 0 4 3.2 58 1 0 0 0 .. ... ... ... ... ... ... 85 2.3 62 1 0 0 1 86 2.9 74 0 0 0 1 87 3.6 71 1 0 0 1 88 3.8 84 1 0 0 1 89 4.3 48 0 0 0 1 [90 rows x 6 columns]
ここではまずこのサンプルデータ全体のカプラン・マイヤー推定量を求めて、プロットすることを考えます。
カプラン・マイヤー推定量を算出する
カプラン・マイヤー推定量を求めるにはKaplanMeierFitterクラスをインスタンス化し、そのfitメソッドに生存期間とイベント発生の有無を指定してフィットさせます。
from lifelines import KaplanMeierFitter
kmf = KaplanMeierFitter()
kmf.fit(durations=larynx_data.time, event_observed=larynx_data.death)
カプラン・マイヤー推定量の値はsurvival_function_属性を用いて取得できます。
print(kmf.survival_function_)
KM_estimate timeline 0.0 1.000000 0.1 0.988889 0.2 0.977778 0.3 0.944444 (中略) 9.3 0.296510 9.6 0.296510 10.1 0.296510 10.7 0.296510
また、生存期間中央値はmedian_survival_time_属性を用います。
print(kmf.median_survival_time_)
6.0
カプラン・マイヤー推定量をプロットする(生存率曲線)
最後にこのカプラン・マイヤー推定量をプロットした生存曲線(カプラン・マイヤー曲線)を描いてみましょう。KaplanMeierFitterクラスのplotメソッドを用いれば簡単にグラフ化することができます。
import matplotlib.pyplot as plt
kmf.plot()
plt.show()
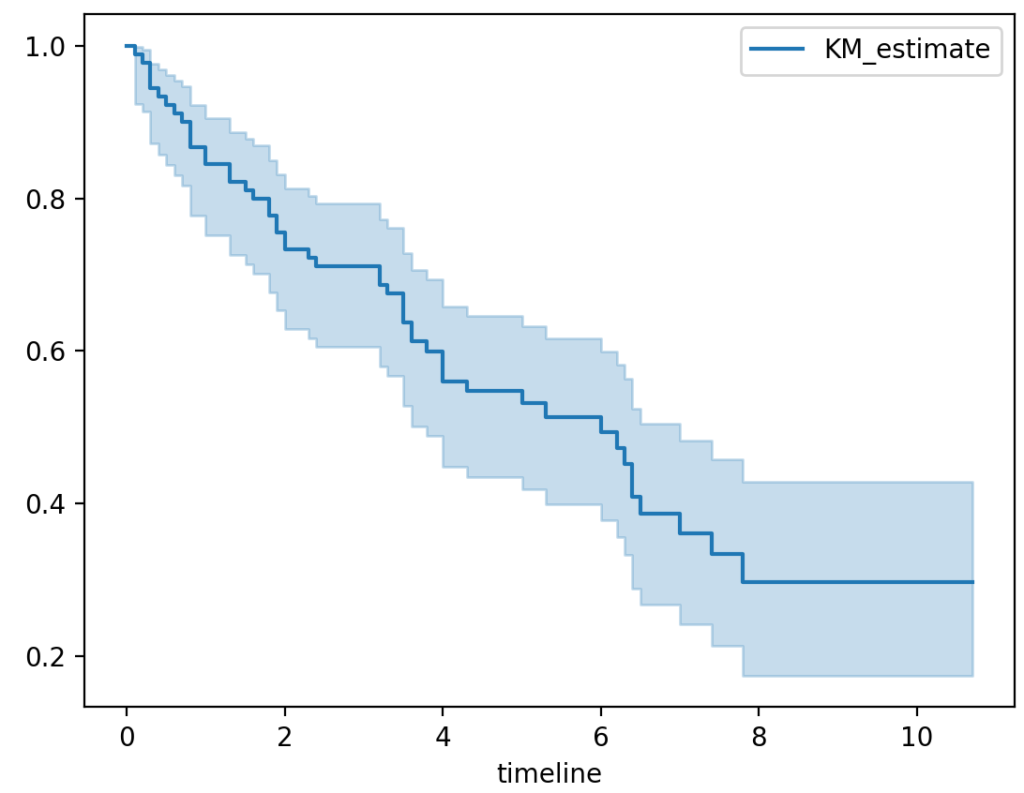
なお、ここで作成されるグラフはmatplotlibのオブジェクトになります。plotメソッドの引数には次のようなものを指定することが可能です。
- show_censors:データの打ち切りを表示するかどうか
- ci_show:信頼区間をグラフに表示かどうか
例えばshow_censorsをTrueとしてデータの打ち切りを打ち切りを「+」で表示し、ci_showをFalseにして信頼区間を消したのグラフは以下のようになります。
kmf.plot(show_censors=True, ci_show=False)
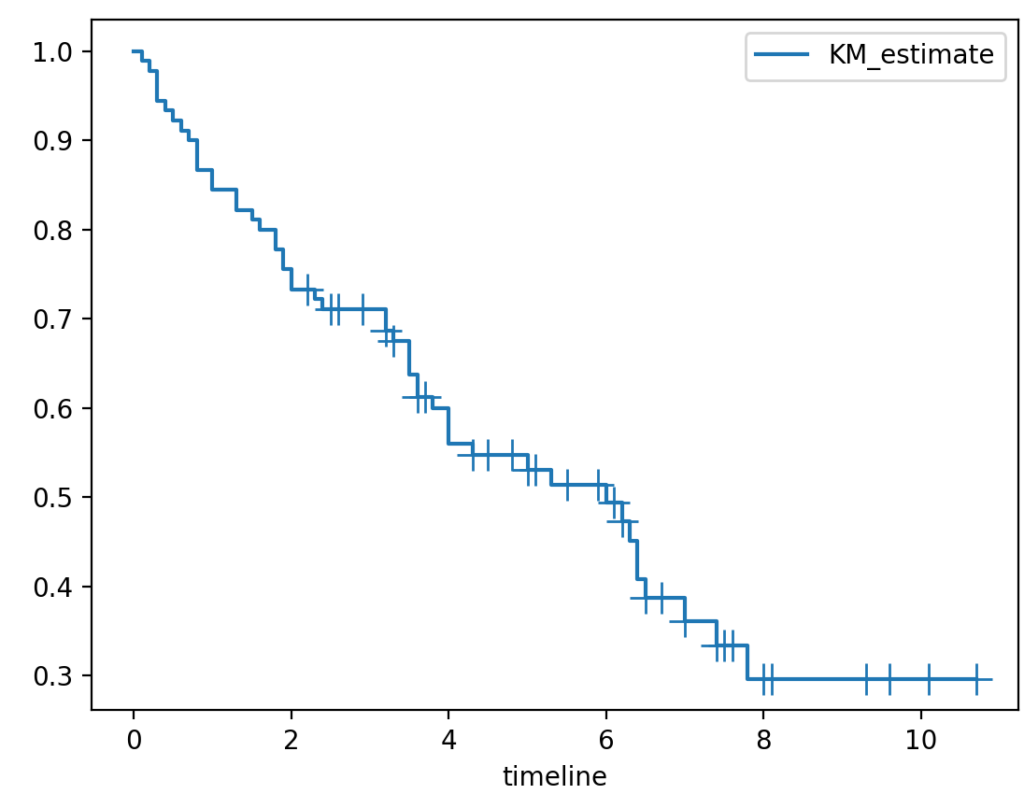
コードまとめ
ここまでのまとめとして、データを読み込んでカプラン・マイヤー曲線を表示させる流れをお示しします。
from lifelines import datasets
from lifelines import KaplanMeierFitter
import matplotlib.pyplot as plt
# データの読み込み
larynx_data = datasets.load_larynx()
# カプラン・マイヤーに当てはめる
kmf = KaplanMeierFitter()
kmf.fit(durations=larynx_data.time, event_observed=larynx_data.death)
# グラフ化する
kmf.plot()
plt.show()
複数のカプラン・マイヤー曲線を重ねて表示させる方法
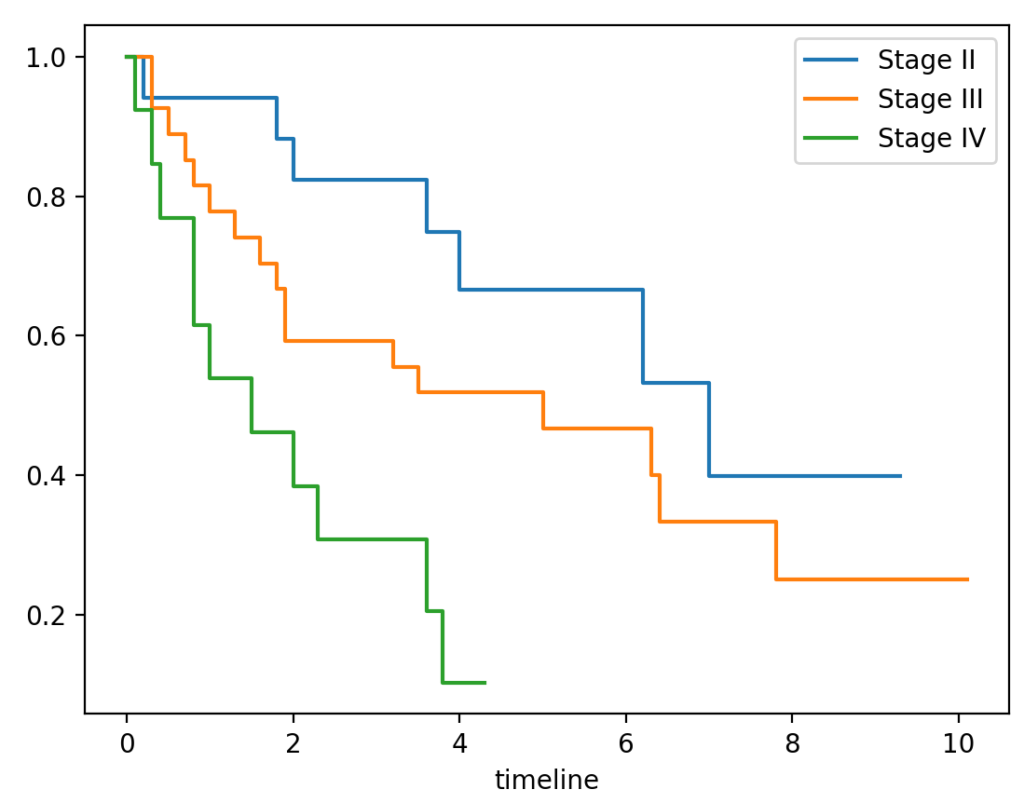
以上は1つのカプラン・マイヤー曲線を表示させる方法でしたが、実際には「異なる治療法を行った2群の生存曲線を比較する」ような場合が多いので、最後に実践編としてその方法をお示しします。
先ほどのデータをStage II、Stage III、Stage IVに分けて、カプラン・マイヤー曲線を表示させてみましょう。KaplanMeierFitterインスタンスごとにplotメソッドを実行するだけでグラフを重ねることができます。その際にfitメソッドでlabelを指定すると、自動的に凡例に表示されます。
from lifelines import datasets
from lifelines import KaplanMeierFitter
import matplotlib.pyplot as plt
# データの読み込み
larynx_data = datasets.load_larynx()
larynx_data_stage2 = larynx_data.query('Stage_II == 1')
larynx_data_stage3 = larynx_data.query('Stage_III == 1')
larynx_data_stage4 = larynx_data.query('Stage_IV == 1')
# カプラン・マイヤーに当てはめる
kmf2 = KaplanMeierFitter()
kmf2.fit(durations=larynx_data_stage2.time, event_observed=larynx_data_stage2.death, label='Stage II')
kmf2.plot(ci_show=False)
kmf3 = KaplanMeierFitter()
kmf3.fit(durations=larynx_data_stage3.time, event_observed=larynx_data_stage3.death, label='Stage III')
kmf3.plot(ci_show=False)
kmf4 = KaplanMeierFitter()
kmf4.fit(durations=larynx_data_stage4.time, event_observed=larynx_data_stage4.death, label='Stage IV')
kmf4.plot(ci_show=False)
# グラフを表示する
plt.show()








コメント