機械学習モジュールのscikit-learnではサンプルデータが用意されており、それをDataFrameとして取り込むこともできます。機械学習のためのサンプルとして使うことはもちろんのこと、DataFrameとして取得すれば様々な解析に用いることができます。ここでは、scikit-learnで提供されているデータセットについて解説を行います。
開発環境
- Python 3.7.7
- scikit-learn 0.23.1

データセットについて
scikit-learnでは、ライブラリに付属している小規模なデータセットと、外部からダウンロードする必要のある大規模なデータセットが提供されています。ここではライブラリに付属している小規模なデータセットの詳細を見てみましょう。
データセットの詳細は公式ドキュメントをご覧ください。
scikit-learnデータセットをDataFrameとして取得する方法
scikit-learnのデータセットはBunchクラスのインスタンスとして取得されます。Bunchクラスのインスタンスは特徴量のデータセットと目標値のデータセットがデフォルトでは共にndarray形式で格納されているので、機械学習の処理には非常に適した形になっていますが、一般的な処理には少し使いづらいかもしれません。そこで、このデータセットをDataFrameとして取得してみましょう。
なお、 Bunchクラスの扱い方については以下の記事をご覧ください。
scikit-learn 0.23 以降
scikit-learn 0.23 以降では、BunchクラスのインスタンスからDataFrameとしてデータを取得するオプションも用意されています。特に、特徴量と目標値のデータセットを統合した形でのDataFrameを取得できるので、機械学習以外の各種解析にも使いやすく便利です。
以下では、アヤメ品種のデータセットを例に説明していきます。最初にデータを取得するときに、load_iris関数でas_frame引数にTrueを指定することで、取得するBunchクラスのインスタンスの特徴量・目標値のデータがndarrayではなくDataFrame / Seriesとなります。さらに、取得したBunchインスタンスのキーにframeを指定することで、特徴量と目標値を統合したDataFrameを取得できます。
from sklearn import datasets
df = datasets.load_iris(as_frame=True).frame
print(df)
scikit-learn 0.22 以前
scikit-learn 0.22 以前では、データセットをDataFrameとして取得するオプションは用意されていません。そのため、データセットで取得したデータから新たにDataFrameを作成する必要がありました。
以下ではボストン住宅価格のデータセットを例として説明していきます。load_boston関数でBunchクラスのインスタンスとしてデータセットを取得した上で、その特徴量部分(説明変数部分)をDataFrameに変換します。そして、最後に目標値部分(目的変数部分)をDataFrameに付け加えます。
from sklearn import datasets
import pandas as pd
# ボストン住宅価格データセットを取得します
boston = datasets.load_boston()
# 取得したデータセットの特徴量部分をDataFrameに変換します
boston_df = pd.DataFrame(boston.data, columns=boston.feature_names)
# データセットの目標値部分をDataFrameに追加します
boston_df['MEDV'] = boston.target
print(boston_df)
データセットの詳細
ボストン住宅価格のデータセット
※ このデータセットは倫理的問題によりscikit-learn 1.0から非推奨となっており、scikit-learn 1.2で削除予定です。
1970年代後半におけるボストンの住宅価格についてのデータセットで、機械学習や統計のサンプルとしてよく用いられるものです。load_boston関数を用いてデータセットを取得します。なお、この関数は非推奨であるためscikit-learn 0.23のアップデートが行われておらず、as_frame引数が用意されていません。
from sklearn import datasets
import pandas as pd
boston = datasets.load_boston()
boston_df = pd.DataFrame(boston.data, columns=boston.feature_names)
boston_df['MEDV'] = boston.target
print(boston_df)
CRIM ZN INDUS CHAS NOX RM AGE DIS RAD TAX PTRATIO B LSTAT MEDV 0 0.00632 18.0 2.31 0.0 0.538 6.575 65.2 4.0900 1.0 296.0 15.3 396.90 4.98 24.0 1 0.02731 0.0 7.07 0.0 0.469 6.421 78.9 4.9671 2.0 242.0 17.8 396.90 9.14 21.6 2 0.02729 0.0 7.07 0.0 0.469 7.185 61.1 4.9671 2.0 242.0 17.8 392.83 4.03 34.7 3 0.03237 0.0 2.18 0.0 0.458 6.998 45.8 6.0622 3.0 222.0 18.7 394.63 2.94 33.4 4 0.06905 0.0 2.18 0.0 0.458 7.147 54.2 6.0622 3.0 222.0 18.7 396.90 5.33 36.2 .. ... ... ... ... ... ... ... ... ... ... ... ... ... ... 501 0.06263 0.0 11.93 0.0 0.573 6.593 69.1 2.4786 1.0 273.0 21.0 391.99 9.67 22.4 502 0.04527 0.0 11.93 0.0 0.573 6.120 76.7 2.2875 1.0 273.0 21.0 396.90 9.08 20.6 503 0.06076 0.0 11.93 0.0 0.573 6.976 91.0 2.1675 1.0 273.0 21.0 396.90 5.64 23.9 504 0.10959 0.0 11.93 0.0 0.573 6.794 89.3 2.3889 1.0 273.0 21.0 393.45 6.48 22.0 505 0.04741 0.0 11.93 0.0 0.573 6.030 80.8 2.5050 1.0 273.0 21.0 396.90 7.88 11.9 [506 rows x 14 columns]
- CRIM : 町ごとの人口一人当たりの犯罪発生率
- ZN : 25,000平方フィートを超える区画に分類される住宅地の割合 (広い家の割合)
- INDUS : 町ごとの非小売業の割合
- CHAS : チャールズ川に接しているかどうか (0 : 接していない、1 : 接している)
- NOX : 一酸化窒素濃度(ppm)
- RM : 1戸当たりの平均部屋数
- AGE : 1940年より前に建てられた持ち家の割合 (古い家の割合)
- DIS : 5つあるボストン雇用センターまでの加重距離
- RAD : 主要高速道路へのアクセス性
- TAX : $10,000 ドルあたりの固定資産税率
- PTRATIO : 町ごとの生徒と先生の比率
- B : 町ごとの黒人の割合
- LSTAT : 低所得者人口の割合
- MEDV : 住宅価格の中央値
アヤメ品種のデータセット
3種類のアヤメのそれぞれ50個体の花びらとガクについて、それぞれその幅と長さの4種類の測定値を記録したデータです。フィッシャーが1936年に発表した論文で使われたアヤメの分類のデータであり、「フィッシャーのアヤメ」として有名なデータセットです。load_iris関数を用いてデータセットを取得します。
from sklearn import datasets
df = datasets.load_iris(as_frame=True).frame
print(df)
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm) target 0 5.1 3.5 1.4 0.2 0 1 4.9 3.0 1.4 0.2 0 2 4.7 3.2 1.3 0.2 0 3 4.6 3.1 1.5 0.2 0 4 5.0 3.6 1.4 0.2 0 .. ... ... ... ... ... 145 6.7 3.0 5.2 2.3 2 146 6.3 2.5 5.0 1.9 2 147 6.5 3.0 5.2 2.0 2 148 6.2 3.4 5.4 2.3 2 149 5.9 3.0 5.1 1.8 2 [150 rows x 5 columns]
- sepal length (cm) : ガクの長さ
- sepal width (cm) : ガクの幅
- petal length (cm) : 花弁の長さ
- petal width (cm) : 花弁の幅
- target : アヤメの種類
このデータセットはseabornの学習用データセットに含まれているものと同じですが、アヤメの種類が機械学習に用いやすいように数字に変換されています。

糖尿病の診療についてのデータセット
糖尿病患者の年齢・性別などの基本情報と採血データから1年後の糖尿病の進行度の関係を表したデータセットです。load_diabetes関数を用いてデータセットを取得します。
from sklearn import datasets
df = datasets.load_diabetes(as_frame=True).frame
print(df)
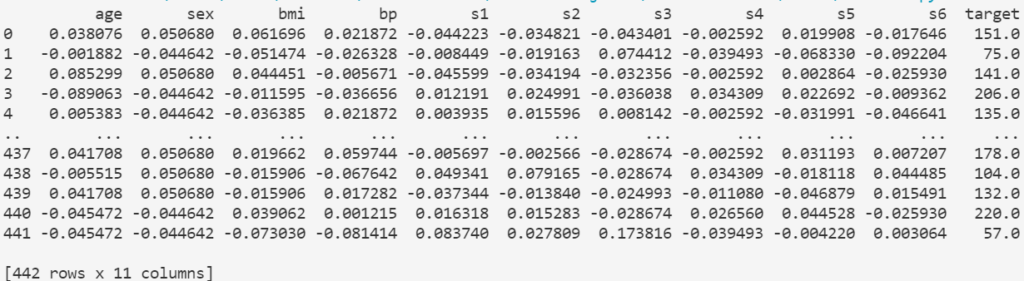
- age : 年齢
- sex : 性別
- bmi : BMI
- bp : 平均血圧
- s1 : 総コレステロール
- s2 : LDL
- s3 : HDL
- s4 : 総コレステロール / HDL
- s5 : トリグリセリド(対数)
- s6 : GLU(血糖値レベル)
- target : ベースラインから1年後の糖尿病の進行度
リファレンスにも以下のように記載されている通り、各々の項目のラベルは実際には不明確なようです。

文献によっては
- s1 : T細胞
- ltg : ラモトリギン
として説明されているものもあります。
手書き文字(数字)の認識に関するデータセット
8 x 8ピクセルの手書き文字(0-9の数字)の画像とその数字を表すデータセットです。load_digits関数を用いてデータセットを取得します。

from sklearn import datasets
df = datasets.load_digits(as_frame=True).frame
print(df)
pixel_0_0 pixel_0_1 pixel_0_2 pixel_0_3 pixel_0_4 pixel_0_5 pixel_0_6 pixel_0_7 pixel_1_0 ... pixel_7_0 pixel_7_1 pixel_7_2 pixel_7_3 pixel_7_4 pixel_7_5 pixel_7_6 pixel_7_7 target 0 0.0 0.0 5.0 13.0 9.0 1.0 0.0 0.0 0.0 ... 0.0 0.0 6.0 13.0 10.0 0.0 0.0 0.0 0 1 0.0 0.0 0.0 12.0 13.0 5.0 0.0 0.0 0.0 ... 0.0 0.0 0.0 11.0 16.0 10.0 0.0 0.0 1 2 0.0 0.0 0.0 4.0 15.0 12.0 0.0 0.0 0.0 ... 0.0 0.0 0.0 3.0 11.0 16.0 9.0 0.0 2 3 0.0 0.0 7.0 15.0 13.0 1.0 0.0 0.0 0.0 ... 0.0 0.0 7.0 13.0 13.0 9.0 0.0 0.0 3 4 0.0 0.0 0.0 1.0 11.0 0.0 0.0 0.0 0.0 ... 0.0 0.0 0.0 2.0 16.0 4.0 0.0 0.0 4 ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... 1792 0.0 0.0 4.0 10.0 13.0 6.0 0.0 0.0 0.0 ... 0.0 0.0 2.0 14.0 15.0 9.0 0.0 0.0 9 1793 0.0 0.0 6.0 16.0 13.0 11.0 1.0 0.0 0.0 ... 0.0 0.0 6.0 16.0 14.0 6.0 0.0 0.0 0 1794 0.0 0.0 1.0 11.0 15.0 1.0 0.0 0.0 0.0 ... 0.0 0.0 2.0 9.0 13.0 6.0 0.0 0.0 8 1795 0.0 0.0 2.0 10.0 7.0 0.0 0.0 0.0 0.0 ... 0.0 0.0 5.0 12.0 16.0 12.0 0.0 0.0 9 1796 0.0 0.0 10.0 14.0 8.0 1.0 0.0 0.0 0.0 ... 0.0 1.0 8.0 12.0 14.0 12.0 1.0 0.0 8 [1797 rows x 65 columns]
ここではDataFrameとして取得しているので、画像データが以下のように対応しています。
- pixel_0_0 : その画像の左上のピクセルの画素値
- pixel_0_1 : その画像の左上の左から2番目のピクセルの画素値
…
- target : その画像が表す数字
このデータセットについてはDataFrameとして取得するメリットはあまりなく、画像データはそのままndarrayとして扱った方がいいかもしれません。
生理学的特徴と運動能力の関係についてのデータセット
体重・ウェスト・心拍などの生理学的特徴と運動能力の関係を表したデータセットです。load_linnerud関数を用いてデータセットを取得します。
from sklearn import datasets
df = datasets.load_linnerud(as_frame=True).frame
print(df)
Chins Situps Jumps Weight Waist Pulse 0 5.0 162.0 60.0 191.0 36.0 50.0 1 2.0 110.0 60.0 189.0 37.0 52.0 2 12.0 101.0 101.0 193.0 38.0 58.0 3 12.0 105.0 37.0 162.0 35.0 62.0 4 13.0 155.0 58.0 189.0 35.0 46.0 5 4.0 101.0 42.0 182.0 36.0 56.0 6 8.0 101.0 38.0 211.0 38.0 56.0 7 6.0 125.0 40.0 167.0 34.0 60.0 8 15.0 200.0 40.0 176.0 31.0 74.0 9 17.0 251.0 250.0 154.0 33.0 56.0 10 17.0 120.0 38.0 169.0 34.0 50.0 11 13.0 210.0 115.0 166.0 33.0 52.0 12 14.0 215.0 105.0 154.0 34.0 64.0 13 1.0 50.0 50.0 247.0 46.0 50.0 14 6.0 70.0 31.0 193.0 36.0 46.0 15 12.0 210.0 120.0 202.0 37.0 62.0 16 4.0 60.0 25.0 176.0 37.0 54.0 17 11.0 230.0 80.0 157.0 32.0 52.0 18 15.0 225.0 73.0 156.0 33.0 54.0 19 2.0 110.0 43.0 138.0 33.0 68.0
- Chins : 懸垂の回数
- Situps : 腹筋の回数
- Jumps : 跳躍の回数
- Weight : 体重
- Waist : ウエスト
- Pulse : 脈拍
ワインの識別についてのデータセット
イタリアの同じ地域の3つの異なるワイナリーで作られたワインの成分を化学的分析に分析した結果です。load_wine関数を用いてデータセットを取得します。
from sklearn import datasets
df = datasets.load_wine(as_frame=True).frame
print(df)
alcohol malic_acid ash alcalinity_of_ash magnesium total_phenols flavanoids nonflavanoid_phenols proanthocyanins color_intensity hue od280/od315_of_diluted_wines proline target 0 14.23 1.71 2.43 15.6 127.0 2.80 3.06 0.28 2.29 5.64 1.04 3.92 1065.0 0 1 13.20 1.78 2.14 11.2 100.0 2.65 2.76 0.26 1.28 4.38 1.05 3.40 1050.0 0 2 13.16 2.36 2.67 18.6 101.0 2.80 3.24 0.30 2.81 5.68 1.03 3.17 1185.0 0 3 14.37 1.95 2.50 16.8 113.0 3.85 3.49 0.24 2.18 7.80 0.86 3.45 1480.0 0 4 13.24 2.59 2.87 21.0 118.0 2.80 2.69 0.39 1.82 4.32 1.04 2.93 735.0 0 .. ... ... ... ... ... ... ... ... ... ... ... ... ... ... 173 13.71 5.65 2.45 20.5 95.0 1.68 0.61 0.52 1.06 7.70 0.64 1.74 740.0 2 174 13.40 3.91 2.48 23.0 102.0 1.80 0.75 0.43 1.41 7.30 0.70 1.56 750.0 2 175 13.27 4.28 2.26 20.0 120.0 1.59 0.69 0.43 1.35 10.20 0.59 1.56 835.0 2 176 13.17 2.59 2.37 20.0 120.0 1.65 0.68 0.53 1.46 9.30 0.60 1.62 840.0 2 177 14.13 4.10 2.74 24.5 96.0 2.05 0.76 0.56 1.35 9.20 0.61 1.60 560.0 2 [178 rows x 14 columns]
- alcohol : アルコール濃度
- malic_acid : リンゴ酸
- ash : 灰分
- alcalinity_of_ash : 灰分のアルカリ性
- magnesium : マグネシウム
- total_phenols : フェノール類全量
- flavanoids : フラボノイド
- nonflavanoid_phenols : 非フラボノイドフェノール類
- proanthocyanins : プロアントシアニジン
- color_intensity : 色彩強度
- hue : 色相
- od280/od315_of_diluted_wines : 希釈ワインのOD280/OD315
- proline : プロリン
- target : ワインの製造元(ワイナリー)
乳がんの診断についてのデータセット
乳房腫瘤に対して実施したFNA(穿刺吸引細胞診)の病理画像と診断結果を表したデータセットです。細胞診の結果は特に細胞核の情報が取得されています。load_breast_cancer関数を用いてデータセットを取得します。
from sklearn import datasets
df = datasets.load_breast_cancer(as_frame=True).frame
print(df)
mean radius mean texture mean perimeter mean area mean smoothness mean compactness mean concavity ... worst smoothness worst compactness worst concavity worst concave points worst symmetry worst fractal dimension target 0 17.99 10.38 122.80 1001.0 0.11840 0.27760 0.30010 ... 0.16220 0.66560 0.7119 0.2654 0.4601 0.11890 0 1 20.57 17.77 132.90 1326.0 0.08474 0.07864 0.08690 ... 0.12380 0.18660 0.2416 0.1860 0.2750 0.08902 0 2 19.69 21.25 130.00 1203.0 0.10960 0.15990 0.19740 ... 0.14440 0.42450 0.4504 0.2430 0.3613 0.08758 0 3 11.42 20.38 77.58 386.1 0.14250 0.28390 0.24140 ... 0.20980 0.86630 0.6869 0.2575 0.6638 0.17300 0 4 20.29 14.34 135.10 1297.0 0.10030 0.13280 0.19800 ... 0.13740 0.20500 0.4000 0.1625 0.2364 0.07678 0 .. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... 564 21.56 22.39 142.00 1479.0 0.11100 0.11590 0.24390 ... 0.14100 0.21130 0.4107 0.2216 0.2060 0.07115 0 565 20.13 28.25 131.20 1261.0 0.09780 0.10340 0.14400 ... 0.11660 0.19220 0.3215 0.1628 0.2572 0.06637 0 566 16.60 28.08 108.30 858.1 0.08455 0.10230 0.09251 ... 0.11390 0.30940 0.3403 0.1418 0.2218 0.07820 0 567 20.60 29.33 140.10 1265.0 0.11780 0.27700 0.35140 ... 0.16500 0.86810 0.9387 0.2650 0.4087 0.12400 0 568 7.76 24.54 47.92 181.0 0.05263 0.04362 0.00000 ... 0.08996 0.06444 0.0000 0.0000 0.2871 0.07039 1 [569 rows x 31 columns]
- mean radius : 細胞核の半径の平均
- mean texture : 細胞核のテクスチャの平均(テクスチャ:グレースケールの画素値の標準偏差)
- mean perimeter : 細胞核の外周長の平均
- mean area : 細胞核の面積の平均
- mean smoothness : 細胞核のなめらかさの平均
- mean compactness : 細胞核のコンパクトさの平均
- mean concavity : 細胞核の輪郭の凹凸の程度の平均
- mean concave points : 細胞核の輪郭の凹凸の個数の平均
- mean symmetry : 細胞核の対称性の平均
- mean fractal dimension : 細胞核のフラクタル次元の平均
- radius error : 細胞核の半径の誤差
- texture error : 細胞核のテクスチャの誤差
- perimeter error : 細胞核の外周長の誤差
- area error : 細胞核の面積の誤差
- smoothness error : 細胞核のなめらかさの誤差
- compactness error : 細胞核のコンパクトさの誤差
- concavity error : 細胞核の輪郭の凹凸の程度の誤差
- concave points error : 細胞核の輪郭の凹凸の個数の誤差
- symmetry error : 細胞核の対称性の誤差
- fractal dimension error : 細胞核のフラクタル次元の誤差
- worst radius : 細胞核の半径の最悪値
- worst texture : 細胞核のテクスチャの最悪値
- worst perimeter : 細胞核の外周長の最悪値
- worst area : 細胞核の面積の最悪値
- worst smoothness : 細胞核のなめらかさの最悪値
- worst compactness : 細胞核のコンパクトさの最悪値
- worst concavity : 細胞核の輪郭の凹凸の程度の最悪値
- worst concave points : 細胞核の輪郭の凹凸の個数の最悪値
- worst symmetry : 細胞核の対称性の最悪値
- worst fractal dimension : 細胞核のフラクタル次元の最悪値
- target : 悪性もしくは良性 (0:悪性、1:良性)









コメント