がんゲノムデータベースの一つであるGDC Data Portal (TCGA)からは様々なゲノムデータを取得することができ、がん研究に携わる人は必ず使いこなさないといけない必須スキルとすら言えます。ここでは、GDC Data Portalの使い方について説明していきます。
GDC Data Portal (TCGA)について
NIH(アメリカ国立衛生研究所)によるがんゲノムプロジェクトで、さまざまながん種について、ゲノムやエピゲノム、トランスクリプトーム、変異情報などのデータを集約して、公開しています。もともと、The Cancer Genome Atlas (TCGA)として広く知られていましたが、現在ではGDC Data Portalの一部として公開されています。
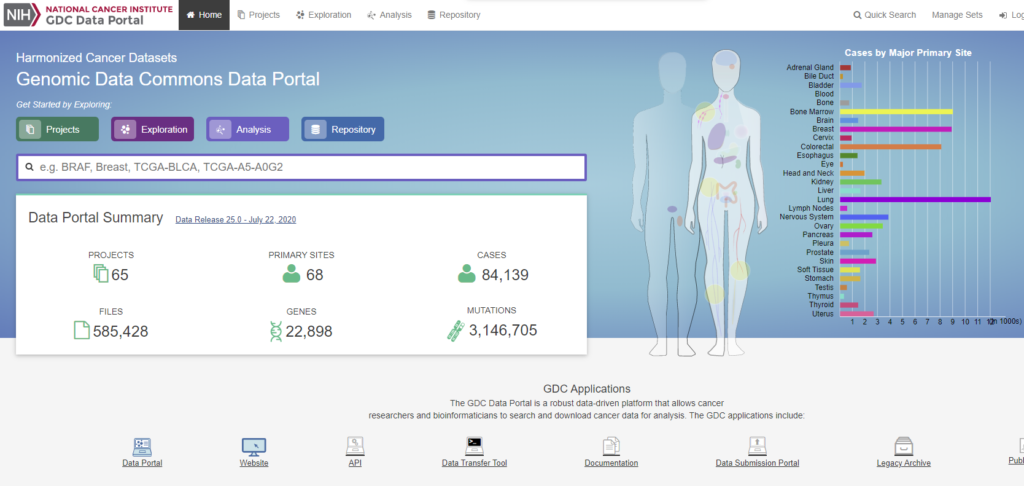
TCGAで公開されているデータには無制限でアクセスできるオープンデータと、アクセス制限がなされた制限付きデータがあります。詳細は公式ドキュメントをご覧ください。
基本的に生データに近いマッピングファイル(***.bam)やバリアントコールファイル(***.vcf)は制限付きデータとして公開されており、RNA-Seqにおける遺伝子発現量を表すFPKM値などの最終的な解析結果がオープンデータとして公開されていることが多いようです。また、基本的な臨床情報(年齢・癌の部位・組織型・ステージ・生死など)もオープンデータとして提供されています。
さらに、TCGAで公開されている情報はいわゆるバイオインフォマティクスの情報だけにとどまらず、病理画像写真もSVS形式でオープンデータとして公開されていたりします。
GDC Data Portal (TCGA)の基本的な使い方
GDC Data Portalの上のタブでデータの表示方法を切り替えます。それぞれ以下のように対応しています。
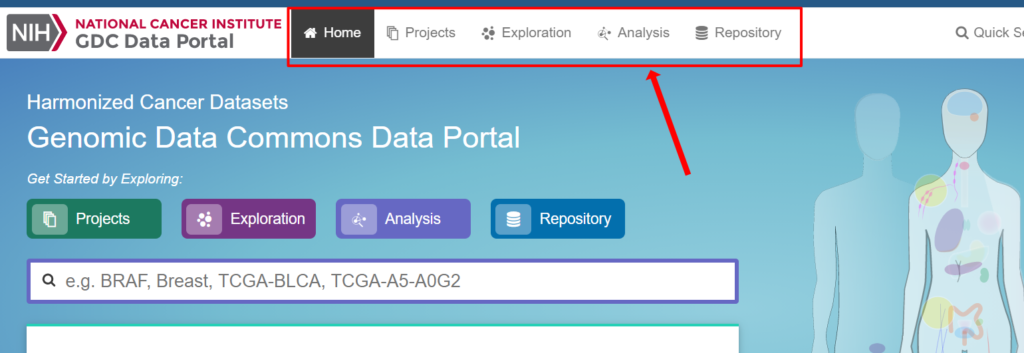
- Projects:プロジェクトごとに閲覧する
- Exploration:症例や遺伝子・変異などデータを探す
- Analysis:自分で新たなデータの検索条件を作成してデータを探す
- Repository:データを表示し、ダウンロードする
「Repository」がデータベース本体で個々のデータがファイルごとに格納されていて、「Project」や「Exploration」「Analysis」で検索条件を指定して分かりやすいように情報を整理して表示させるイメージです。
例1:乳がんの症例を探してみる
上のタブを「Exploration」にして、左側の条件設定欄で「breast」にチェックを入れてみましょう。
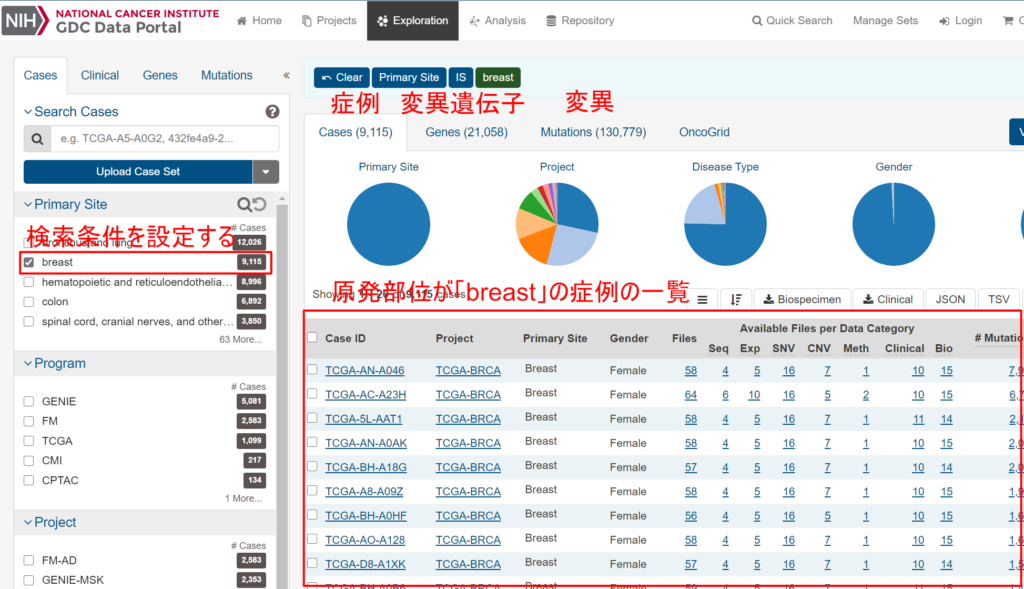
これで原発部位が「breast」の症例の一覧が表示されました。変異遺伝子の一覧やその頻度などは「Genes」を、実際の変異形式の一覧やその頻度などは「Mutations」をクリックしてください。
ここでは、先ほどの症例一覧の一番上にあるCase ID「TCGA-AN-A046」のデータを参照してみましょう。以下のようにこの症例に関連づいたデータが以下のように表示され、合計で58個のファイルがあることが分かりました。「Summary」の中の「images」は病理組織画像を表しており、実際のHE染色の画像を閲覧することも可能です。他には、RNA-Seqのデータもあり遺伝子発現定量データも取得することができます。
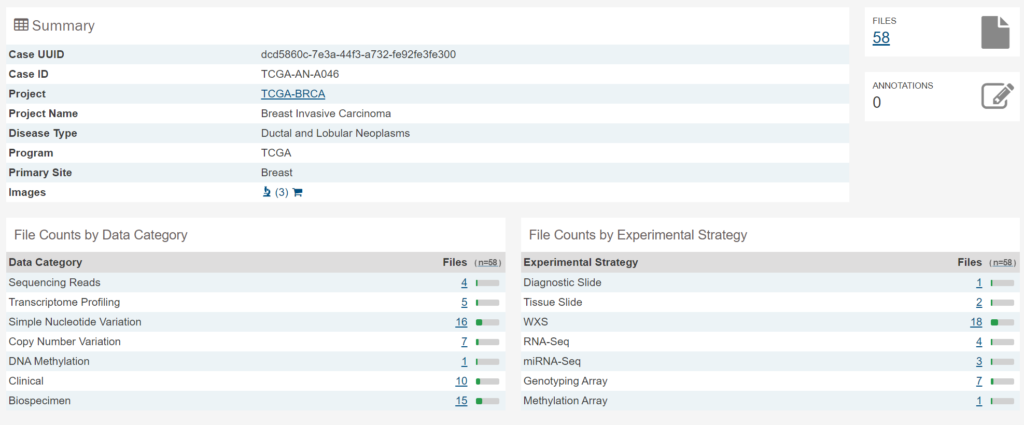
さらにもう少し下の方を見てみると以下のような情報が得られます。例えばこの症例の場合は診断時の年齢が68歳で、組織型は「Infiltrating duct carcinoma」、病期は「IIA期」であったことが分かります。また、「Demographic」では人種・性別・生死などの情報も得られます。
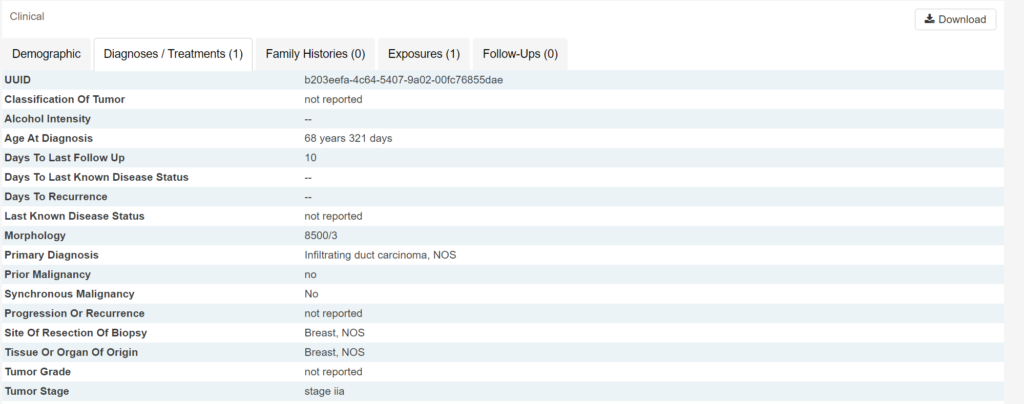
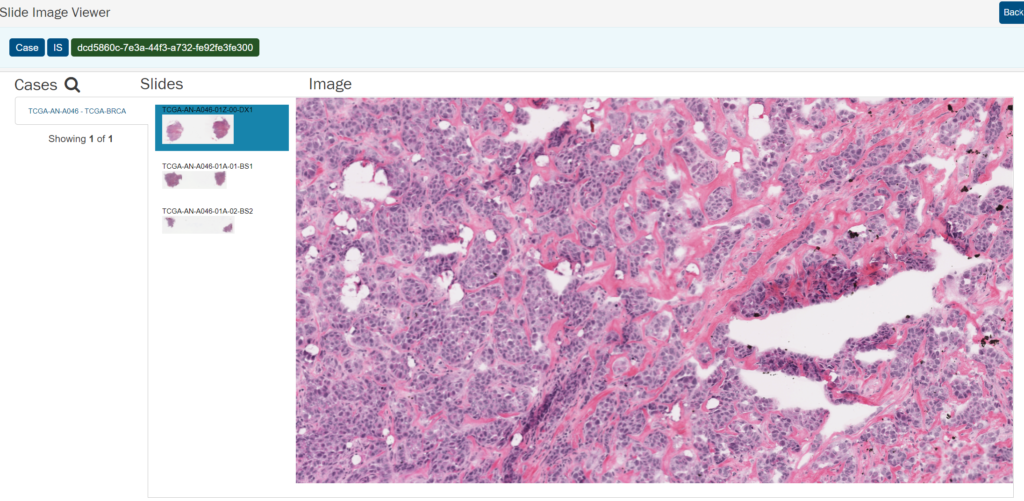
実際の組織画像も確認してみましょう。「Summary」の「images」の顕微鏡のアイコンをクリックすると以下のようにHE画像を閲覧できます。「Summary」の「images」のショッピングカートのアイコンをクリックすればダウンロードすることも可能です。
例2:全乳がん症例の遺伝子発現定量値を取得する
ここでは遺伝子発現量の定量値としてFPKM値を求めてみましょう。ちなみにGDC Data Portalで取得できる遺伝子発現量は以下のようにHTSeqを用いてカウントされたものが主体となります。

それでは、上のタブを「Repository」にして、左側の条件指定欄で以下のように「RNA-Seq」「HTSeq – FPKM」「breast」にチェックを付けてみましょう。
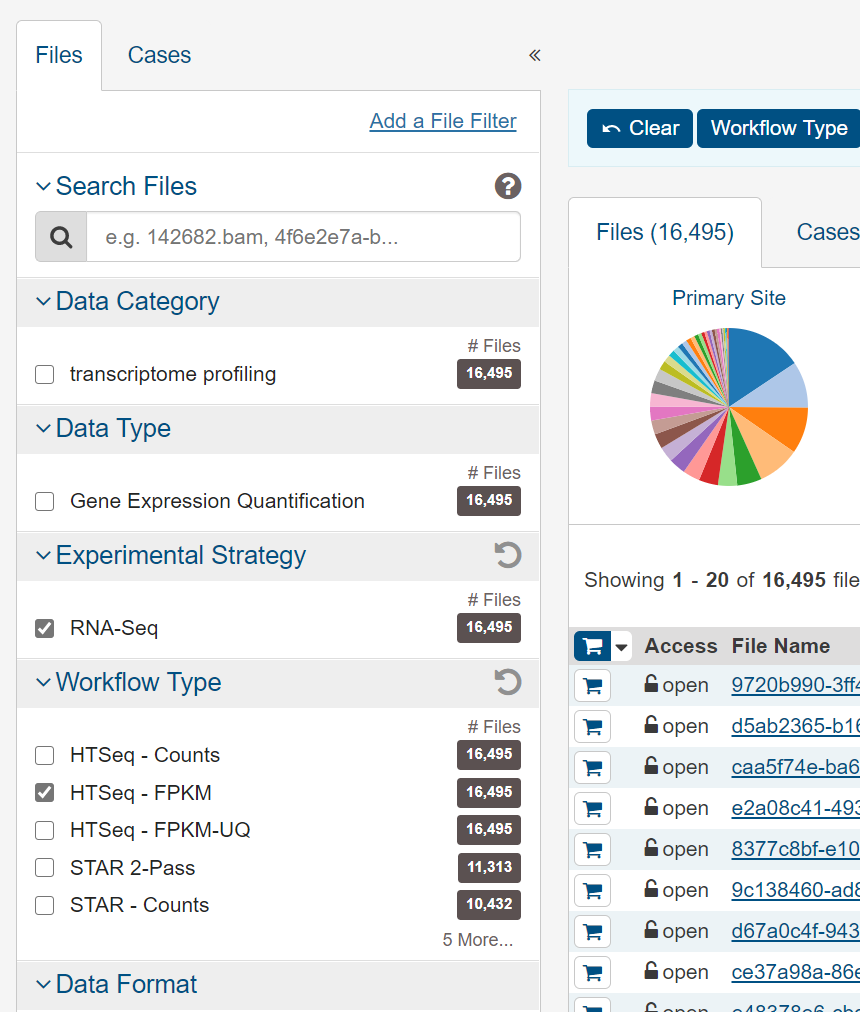
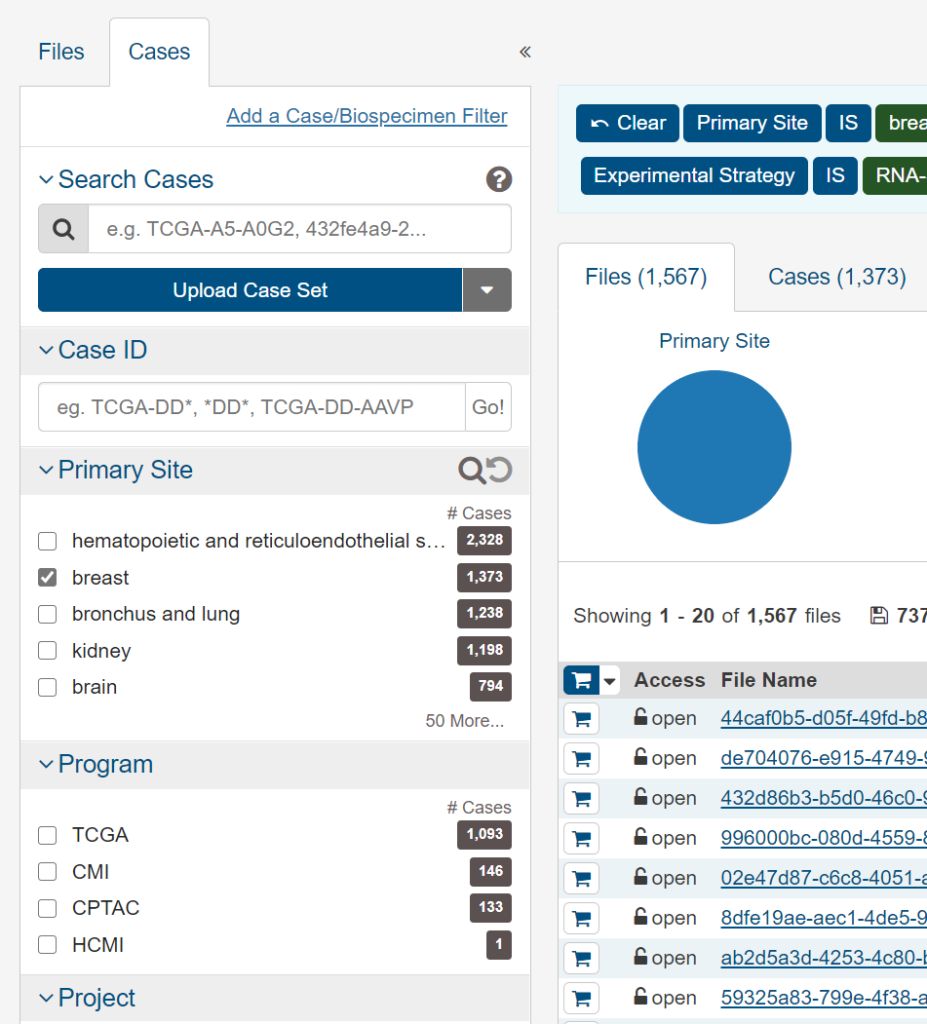
これで乳がん1567症例(1373人)のFPKM値が抽出されました。ダウンロードするためにはまずこれらをカートに入れる必要があるので、「Add All Files to Cart」をクリックします。
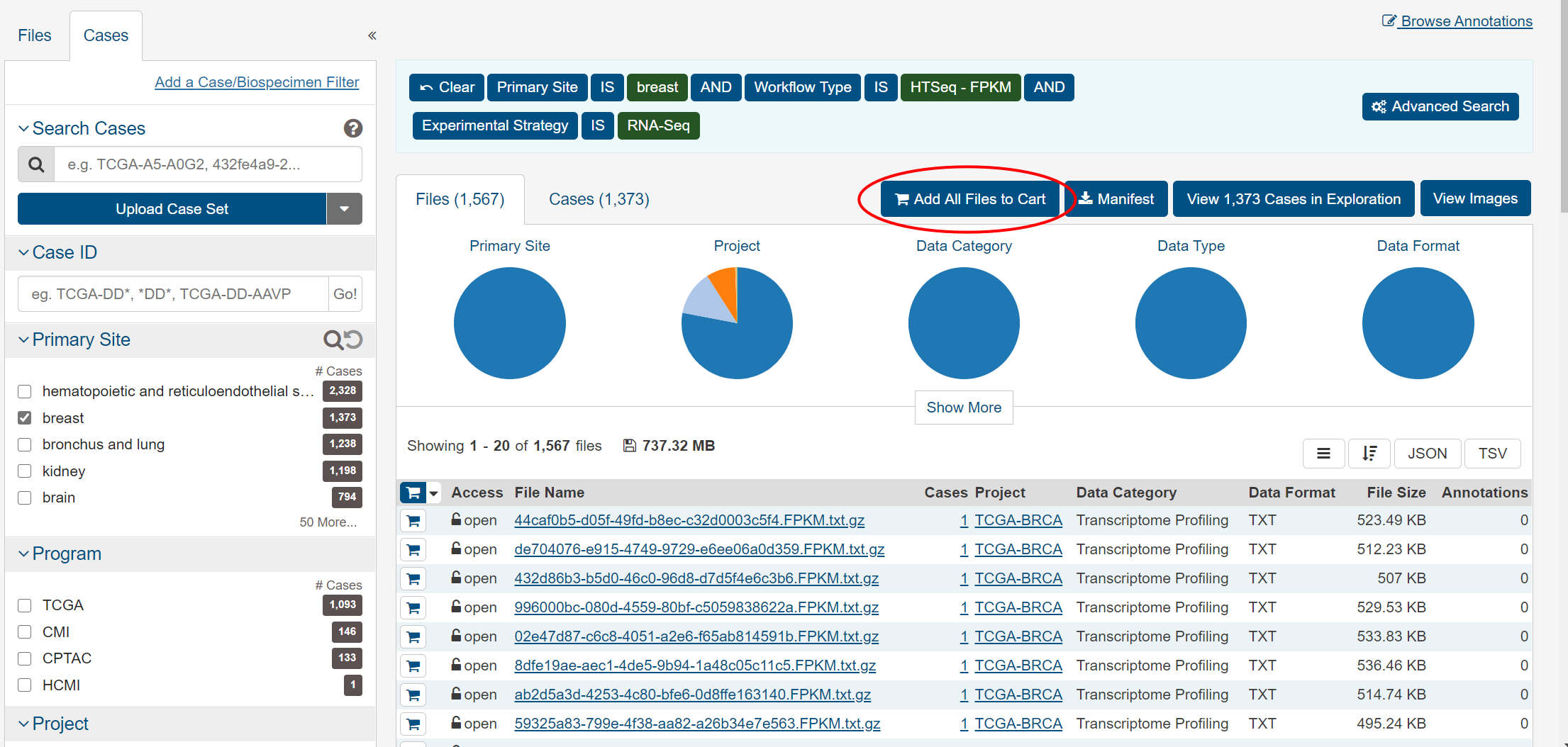
必要なファイルだけ選んで取得する場合は、それぞれのファイルの左側のショッピングカートマークのボタンをクリックして個別にカートに追加してください。取得したいファイルをカートに追加したら、右上の「Cart」をクリックしてカートを表示させて、「Download」から「Cart」を選択してダウンロードを実行します。
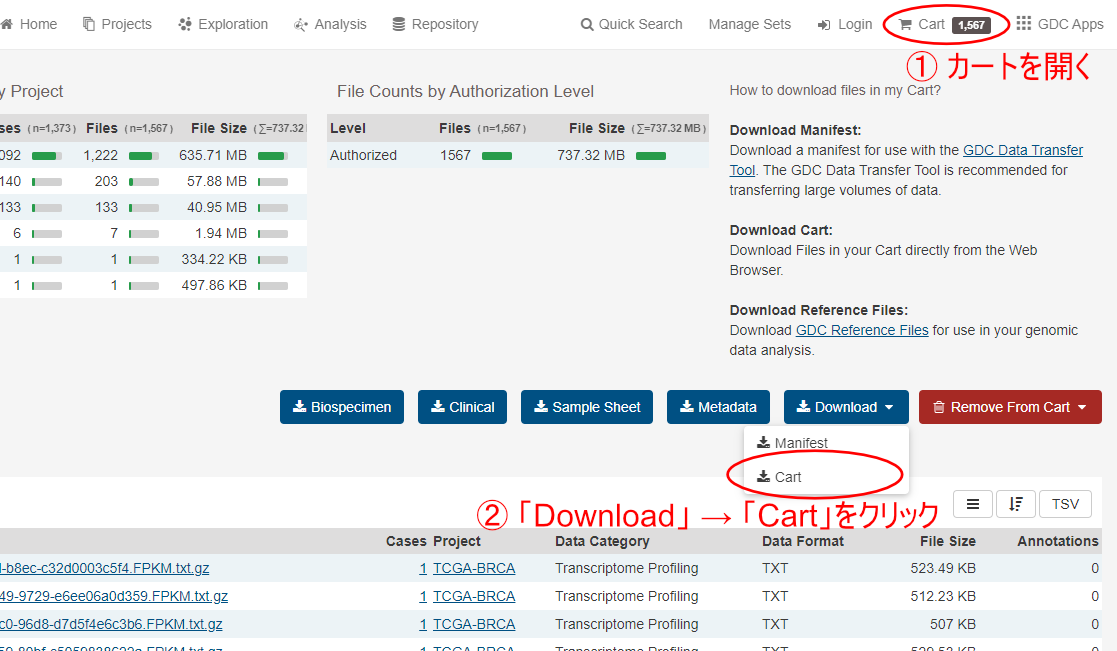
なお、一つずつファイルをダウンロードする場合はカートに入れる必要はなく、ダウンロードしたいファイルを選択して、「Download」ボタンをクリックして下さい。
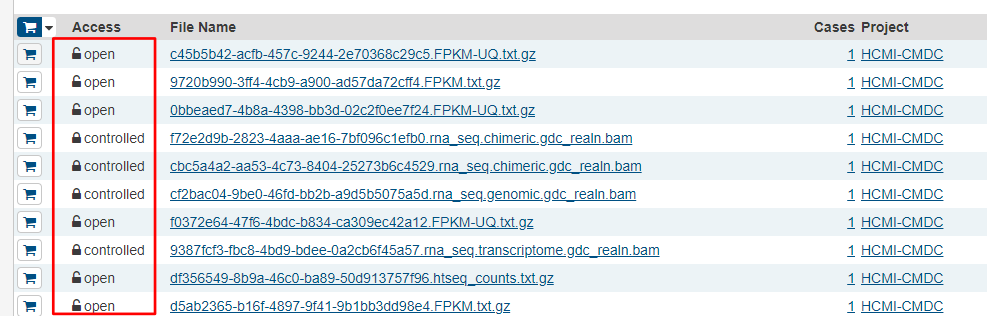
また今回の例で用いたFPKM値のデータはすべてオープンデータとして公開されているものですが、以下のようにAccessがControlledになっているものは制限付きデータであり、取得するには特別の申請が必要になります。
なお、ここで取得できるデータはFPKM(RPKM)値ですが、遺伝子発現量の比較において現在はTPM値が用いられています。FPKM(RPKM)値をTPM値に変換する方法については以下の記事をご覧ください。

カートの便利な使い方
カートに入れたデータに付随する情報や臨床データを取得する
先ほどの例ではRNA-Seqの結果として得られた乳がんの遺伝子発現の定量値を取得しましたが、それぞれのRNA-Seqの結果はどのような症例のものだったのかが知りたくなる場合があります。例えば、「このような遺伝子発現が亢進している場合は予後がどうなるのか」「このような遺伝子発現パターンの場合は腫瘍の進行にどのような影響を及ぼすのか」などといったことを調べるためにはRNA-Seqの結果と臨床データとを組み合わせて解析する必要があります。
そのような場合は、ダウンロードしたいデータをカートに入れたら、以下の「Biospecimen」「Clinical」「Sample Sheet」「Metadata」からそのデータに関連づいた臨床情報やその他の情報を取得してみましょう。
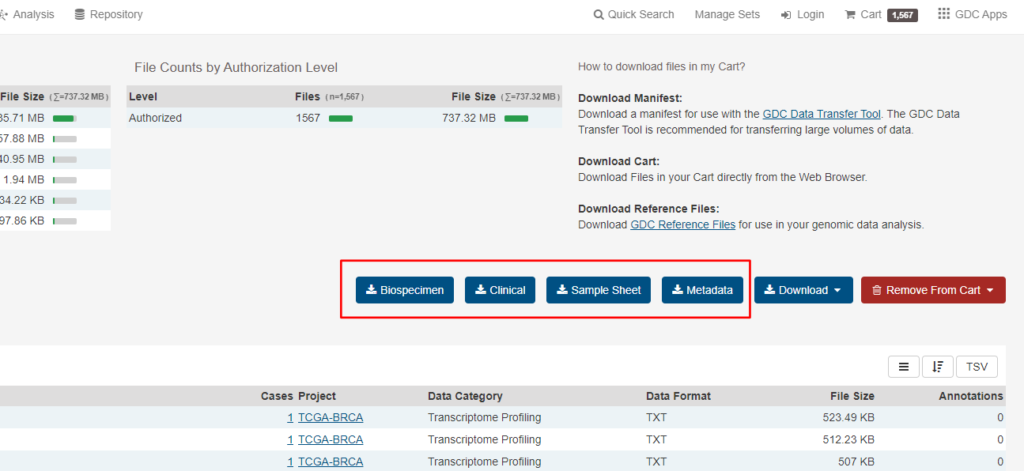
- Biospecimen:検体(サンプル)についての情報
- Clinical:臨床情報
- Sample Sheet:カート内のファイルと、データや検体(サンプル)のIDとを関連付けるための一覧
- Metadata:カート内のファイルについてのメタ情報
GDC Data Transfer Toolを用いてダウンロードする
カートに入れたデータをブラウザから直接ダウンロードすることもできるのですが、大量のデータがある場合はGDC Data Transfer Toolという専用のデータ転送ツールを使用することが推奨されています。GDC Data Transfer Toolにはコマンドライン版とGUI版があるので、直感的に分かりやすいGUI版をインストールしてみましょう。
※ GDC Data Transfer ToolのGUI版は現在β版となっているので、ダウンロードに何か不具合のある場合はコマンドライン版を使用してください。
GDC Data Transfer Toolのページから「Downloading the GDC Data Transfer Tool User Interface (Beta)」の項目の自分の環境に合ったプログラムをダウンロードします。

ここでは、Windows版の「dtt-ui_v0.6.0_Windows_x64.zip」をダウンロードしましょう。zipファイルを解凍し、インストーラを実行してプログラムをインストールします。インストールが終了すると設定画面が表示されますが、基本的に初期設定で問題ありません。(ダウンロードファイルの保存場所は適宜変更してください)
GDC Data Transfer Toolを使用する場合も、まずはダウンロードしたいデータをカートに入れるところまではブラウザからのダウンロードと同じです。カートにダウンロードしたいデータを入れたら、「Download」→「Manifest」を選択し、データ本体ではなくダウンロードするデータのリストを取得します。
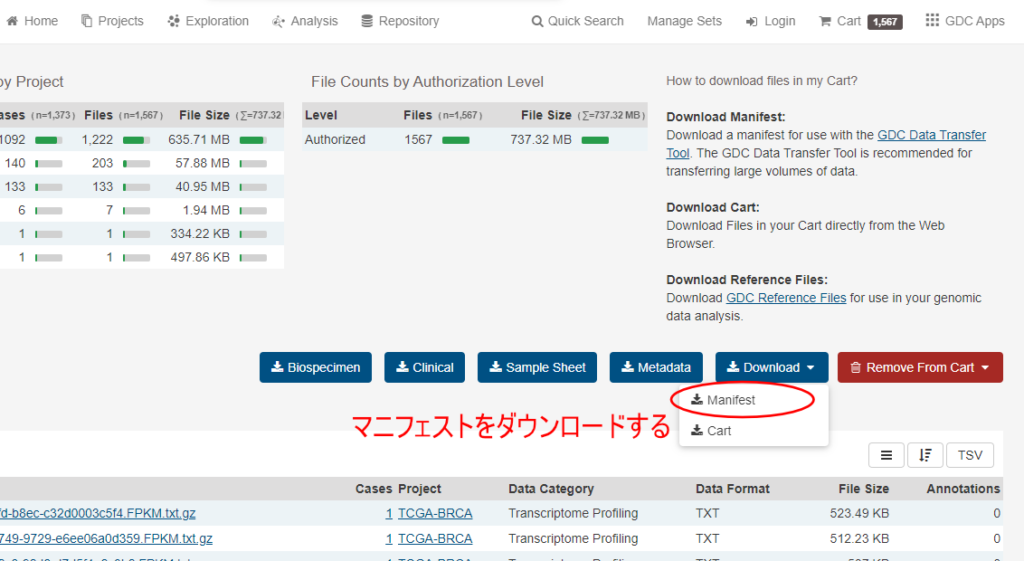
続いて、今ダウンロードしたマニフェストをGDC Data Transfer Toolに指定することで、ダウンロードを実行できます。
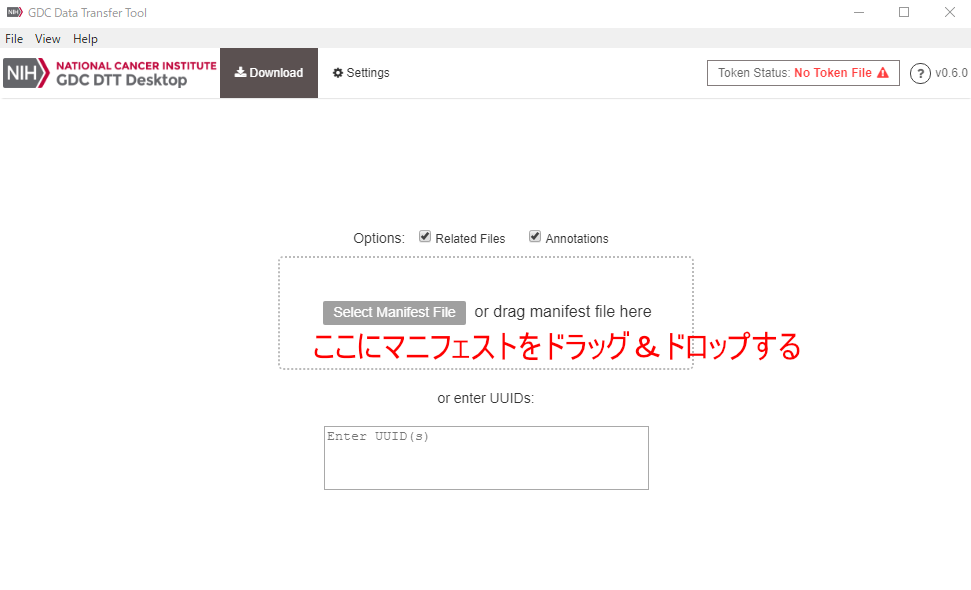
GDC Data Transfer Tool (GUI版) の詳しい使い方は公式ドキュメントをご参照ください。
取得したデータの解凍方法
GDC Data Portalのデータはgzip形式やtar形式などのLinuxで一般的な圧縮フォーマットで構成されているので、Windowsで扱う際は専用の圧縮・解凍ソフトが必要になります。例えば7-zipなどの圧縮・解凍アプリを用いて解凍してください。
※ Microsoft Storeに登録されているのは公式版ではありませんが、GNU LGPLのライセンスのもとでオープンソースで公開されています。








コメント