バイオインフォマティクスの解析はLinuxやそれをベースとしたmacを用いて行われることが一般的ですが、圧倒的に多くの人は普段Windowsを使っていますよね?「Windowsでゲノム解析ができないの?」と一度は皆さん思ったのではないでしょうか?
このサイトではWindowsユーザーがWindowsのマシンを用いてゲノム解析を行う方法を解説します。
目指すべきゲノム解析環境の概要
残念ながらバイオインフォマティクスの領域においてはWindowsでは動作しないアプリケーションも多くあり、すべてをWindowsで完結させるのは不可能なのですが、ユーザー側としては通常のWindowsのアプリケーションを操作しているのとまったく同じような環境を目指します。
- すべての処理をWindowsアプリケーションの中で完結させる
- ゲノム解析で用いたファイルはすべてWindowsで管理する
- スクリプトはPythonを使う(Linuxのシェルスクリプトは用いない)
Windowsユーザーにはなじみの薄いLinuxのシェルスクリプトは用いないで、その代わりPythonを使っています。Pythonはデータサイエンス分野をはじめとして様々な分野に応用可能な万能言語なので、詳しくない方はこれを機会にPythonに慣れてみるといろいろな可能性が広がると思います!
個人的にはLinuxのコマンドも完全に排除して、プラットフォームに依存しない100% Pythonでコマンドを書きたいですが、やはりLinuxコマンドを用いた方が簡単なことも多いので、そこはある程度妥協しています。(できる限り代替となるPythonコマンドも併記するようにします)
WSLの導入
バイオインフォマティクス分野ではWindowsに対応していないアプリケーションもあり、Linuxがデファクトスタンダードとなっています。そこで、WindowsにもLinux環境を導入してみましょう。Windows10にはWindows Subsystem for Linux (WSL)という機能が備わっており、それを有効化するだけで誰でもLinux環境を無料で簡単に構築することができます。
WSLを使うのに必要なのは次の2つのプロセスのみです。
- 「Windowsの機能の有効化または無効化」からWindows Subsystem for Linuxを有効化する
- Microsoftストアから好みのLinuxをインストールする(ここではUbuntuをインストールします)
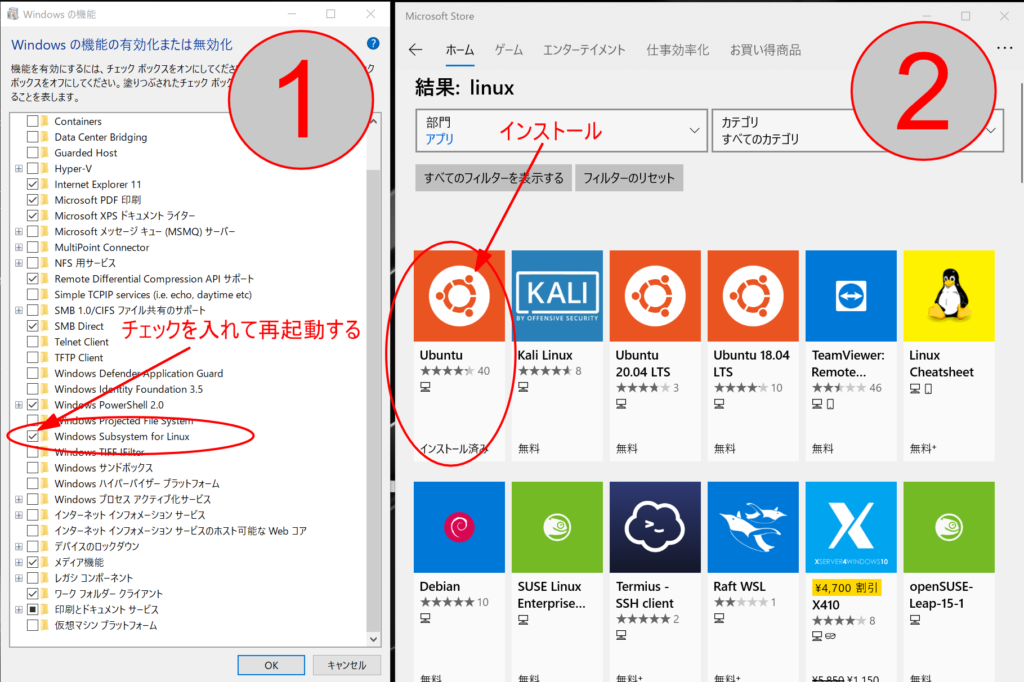
WSLの導入方法はQiitaに記事を書きましたので、こちらをご覧ください。
WSL(ここではUbuntu)を導入したらまず一度起動してみてください。初回起動時にユーザー名とパスワードを設定するように要求されるので、指示に従ってユーザー名とパスワードを設定します。
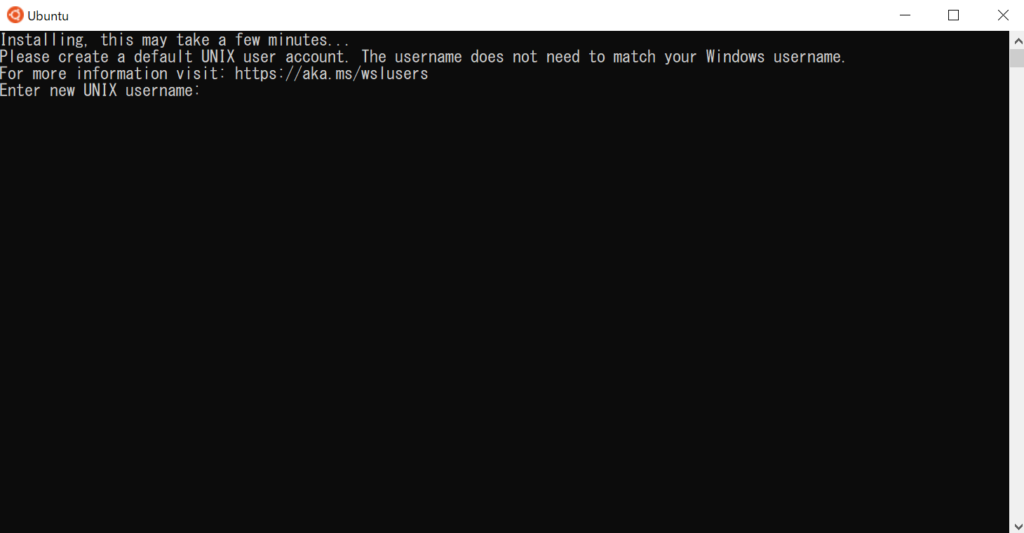
(パスワードの設定の際に、キーボードで入力してもWindowsのように「●●●●」などの文字列は表示されませんが、ちゃんと入力できているのでそのまま続けてください)
さらに、以下のコマンドを入力してインストールしたUbuntuを最新の状態にアップデートしておきましょう。
sudo apt update
Visual Studio CodeからWSLにリモート接続する
Linuxの導入ができましたが、結局Windowsの中のLinuxのコンソールを立ち上げてLinuxコマンドを入力していくことになります。また、LinuxのホームディレクトリにはWindows側からアクセスできないので、ファイルの保存先もLinuxとWindowsとは別々になってしまっています。これでは「Windowsで解析している」とは言えませんよね。
そこで、ここではVisual Studio Codeを導入して、Visual Studio CodeからWSLにリモート接続する環境を構築します。
Visual Studio Codeの導入
Visual Studio CodeはMicrosoftのオープンソースのテキストエディタ―です。まずはVisual Studio Codeのインストールをしましょう。
Visual Studio Codeの拡張機能のインストール(ローカル環境)
次に、Visual Studio Codeの拡張機能でWSLにリモート接続するためのツールを導入しましょう。
WSLへのリモート接続
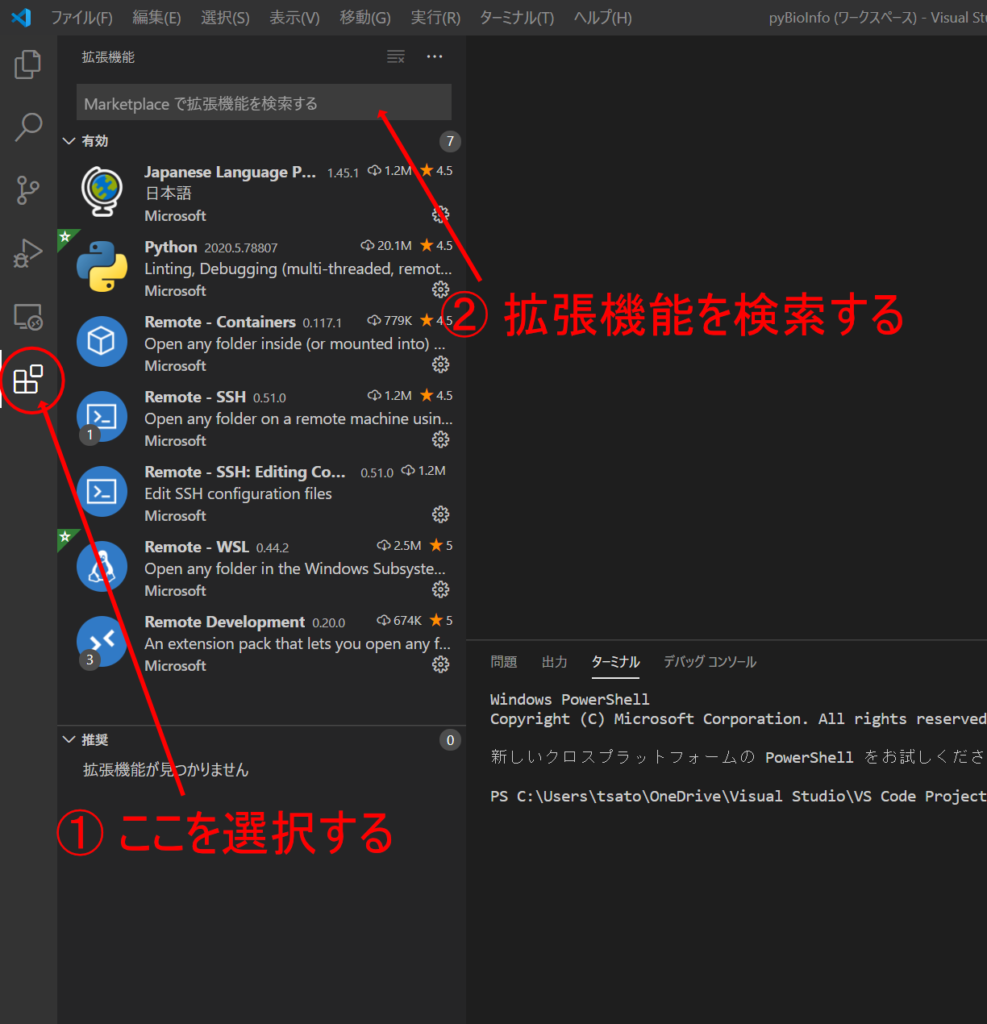
Visual Studio CodeのWSLへのリモート接続に必要な拡張機能をインストールします。左側のメニューから拡張機能を選択して、検索ボックスから拡張機能を検索してインストールしてください。
Visual Studio Codeの日本語化(任意)
Visual Studio Codeの日本語化は必須ではありませんが、この拡張機能をインストールすることで日本語化ができます。
※ ドキュメントには「インストールして設定が必要」と書いてありますが、自分の環境ではインストールして再起動するだけで日本語化されました。
Visual Studio CodeからWSLに接続する
Visual Studio Codeの左下の >< のようなアイコンをクリックしてWSLにリモート接続できます。
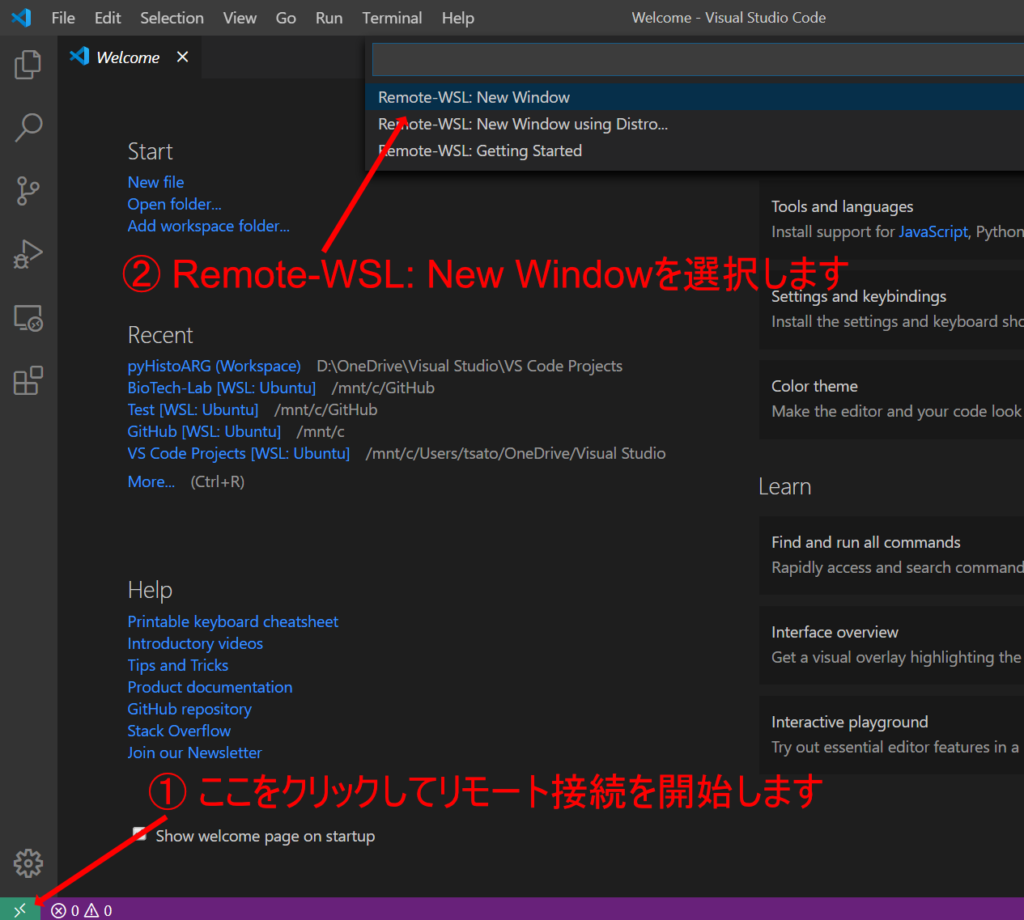
WSLに接続すると左下のアイコンがこのように変わります。
以下の作業は、Visual Studio CodeからWSLに接続した状態で行ってください。
ゲノム解析に必要なツールの導入
パッケージ管理システム(conda)の導入
Ubuntuにインストールするアプリケーションを自分ですべて管理することは困難なので、何らかのパッケージ管理システムを使用することをお勧めします。パッケージ管理システムを使用することで、どのアプリのどのバージョンを、どこのフォルダにインストールしたかや、インストールしたアプリケーションの依存関係についても意識することなく、コマンド一つで簡単にインストールすることができるようになります。
ここでは、一般的に使われていて、バイオインフォマティクスツールも広くカバーしているパッケージ管理システムとしてcondaを用います。
condaはAnacondaかMinicondaのどちらかで導入するのが一般的です。
- Anaconda:conda + Python + データサイエンスに用いる多くのパッケージ
- Miniconda:conda + Python + 最小限のパッケージ
MinicondaはUbuntuのターミナルに以下のコマンドを入力してインストールできます。
curl -O https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
sh Miniconda3-latest-Linux-x86_64.sh
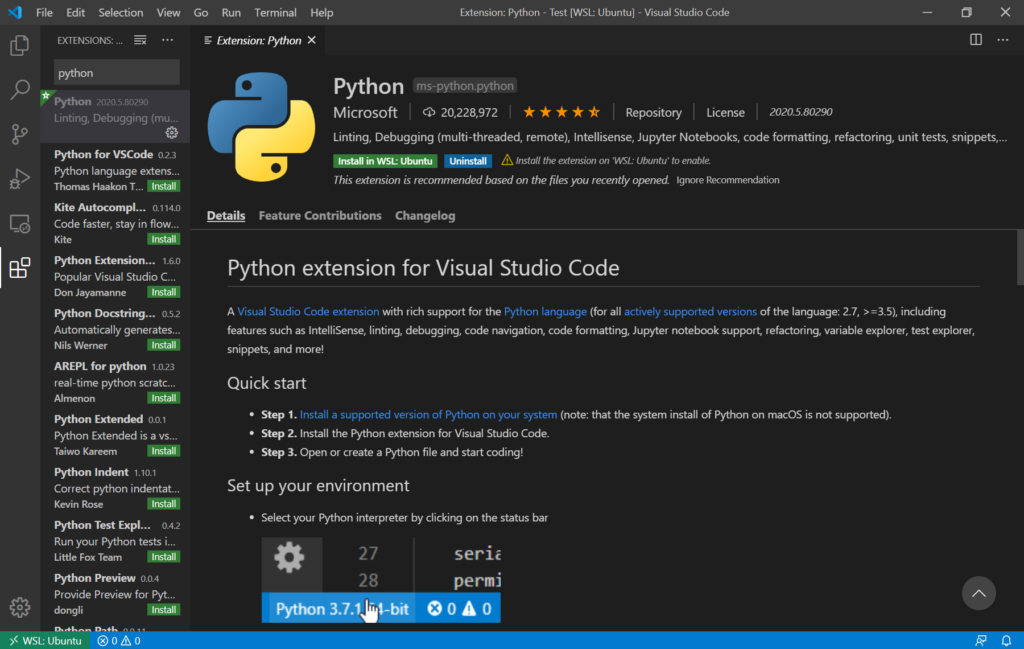
Python 拡張機能の導入
Pythonの開発にはPython拡張機能のインストールが必要になります。なお、Visual Studio Codeの拡張機能はアプリ全体にかかわるものはローカルでもリモート接続でも共有されますが、それ以外のものはそれぞれの環境ごとにインストールする必要があります。Python拡張機能をすでにインストール済みであっても、リモートのWSL環境上に新たにインストールする必要がありますので注意しましょう。
ここでは、Pythonの開発が必要になりますので、WSL環境にリモート接続した状態でリモート環境にPython拡張機能をインストールしてください。
作業ディレクトリの設定とこれまでの設定の保存
デフォルトでは作業ディレクトリはUbuntuのホームディレクトリになっていますが、Windowsからアクセスできないので作業ディレクトリを変更しておきましょう。
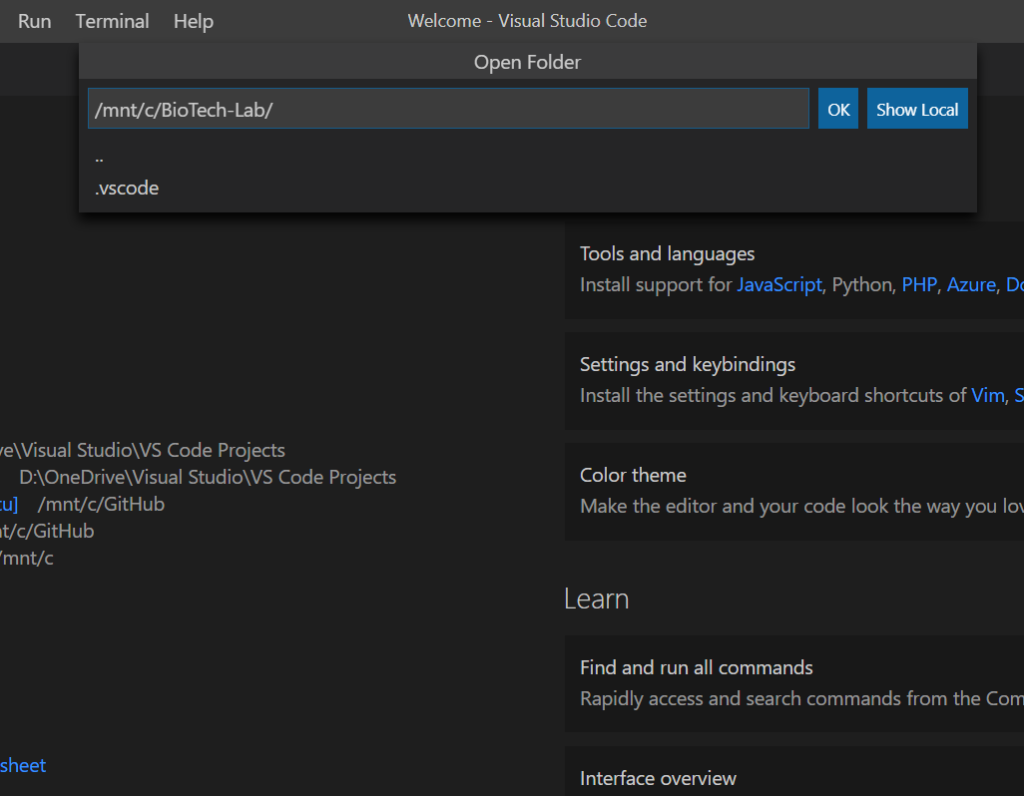
C:\BioTech-LabというフォルダにUbuntu側からアクセスするときは、/mnt/c/BioTech-Labとなります。
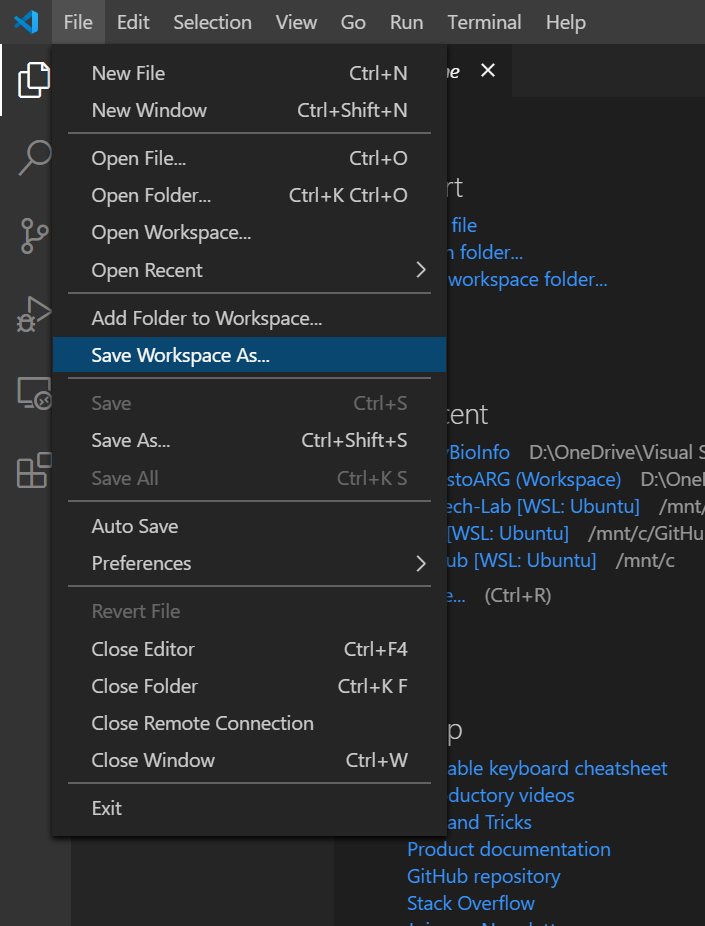
これでUbuntuターミナルの作業ディレクトリが指定したフォルダに変更されました。最後に以上までの設定を保存しましょう。
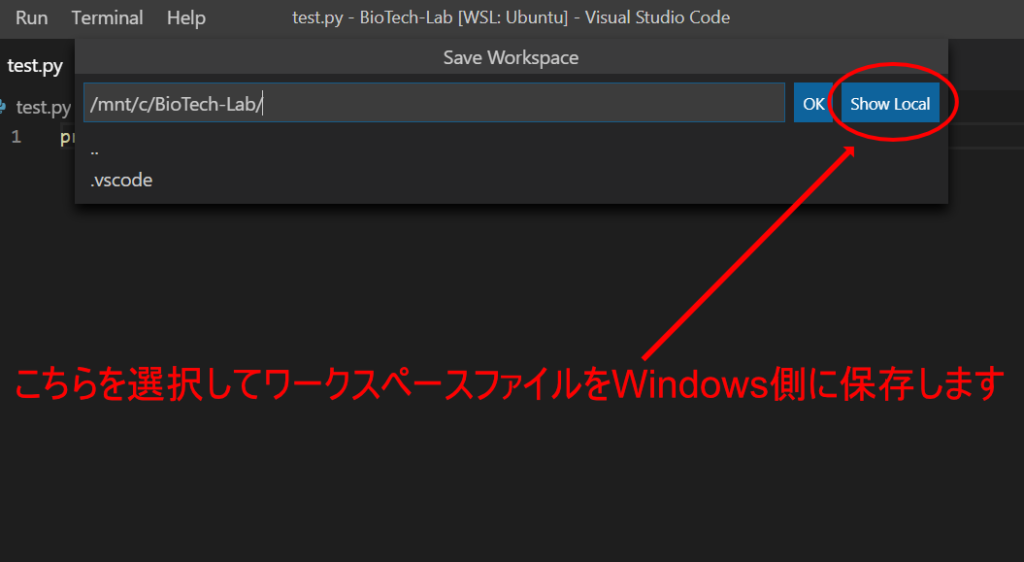
ここでのポイントは、ワークスペースファイルの保存はShow Local (ローカルを表示します)でダイアログを表示させて、Windowsのファイルシステムに保存することです。それにより「WSLにリモート接続する」という設定も保存されますが、/mnt/c/BioTech-Lab/***のようなUbuntu側のディレクトリに保存してしまうと、ワークスペースが保存場所からの相対パスで保存されるので、「WSLにリモート接続する」という設定が保存されません。そのほか、Jupyter Notebookが正しく読み込めなかったりといろいろ問題があるようです。
これで次回からはこのワークスペースを開けば、この状態で開くことができるようになります。
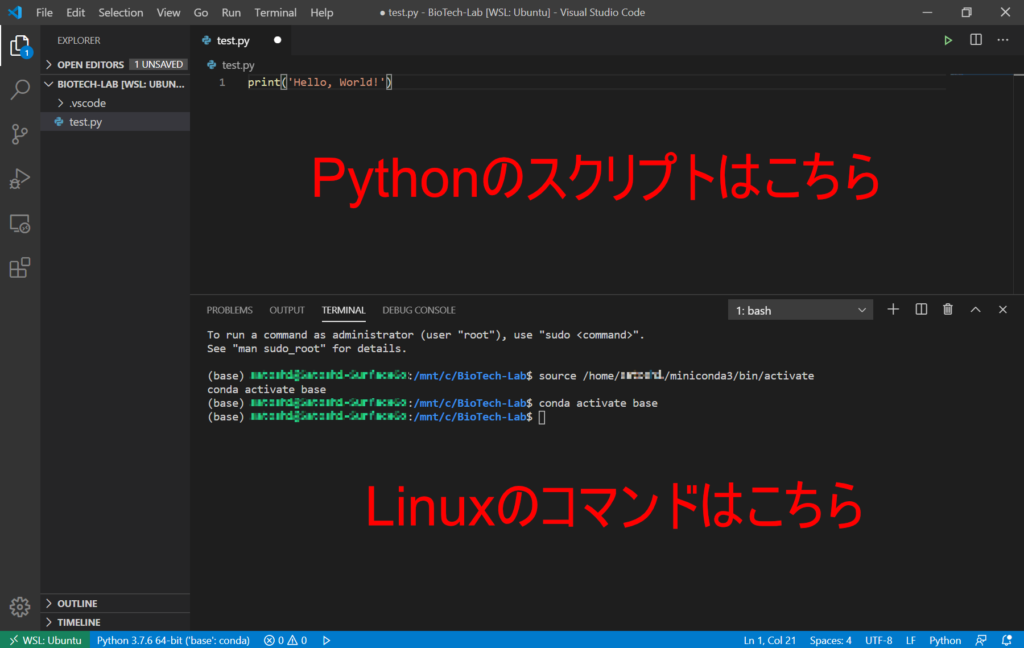
最後に、「Hello, World !」
Python スクリプトファイルの実行
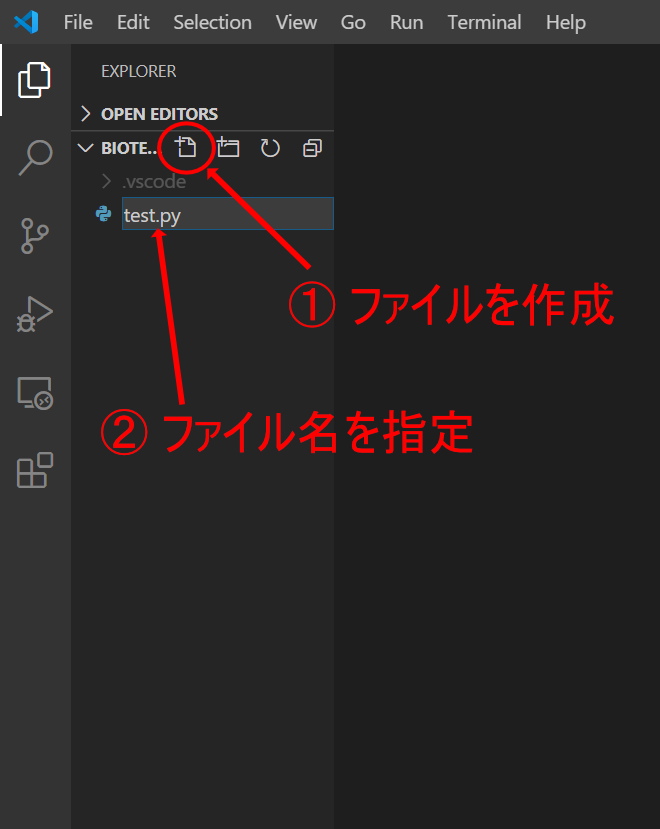
左側のエクスプローラで「*****.py」ファイルを作成し、そこにPythonのコーディングをしていきます。
F5キーでPythonファイルを実行してみましょう。Pythonインタープリターの選択画面が出たら、conda環境のPythonを選びましょう。
UbuntuのターミナルにHello, World!が表示されたら、Ubuntuの中のPythonプログラムを起動することができたということです。
注意:必ずconda環境のPythonで実行していることを確認してください
初回のPython起動時にPythonインタープリターの選択画面が出ますが、「Python 3.〇.〇 64-bit (‘base’:conda)」となっているconda環境でインストールしたPythonを選択してください。
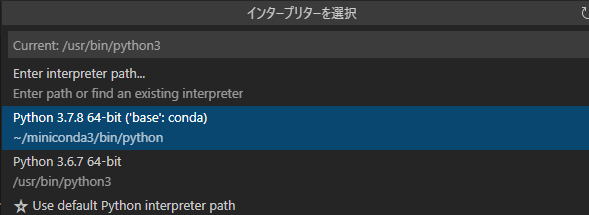
UbuntuにはデフォルトでPythonがインストールされているので、そちらで実行してしまっているとせっかくconda環境で新たなライブラリを導入しても反映されなくなってしまいます。
もし導入したライブラリが反映されていない場合はVisual Studio Codeの左下のPythonインタープリターの部分を確認して、conda環境で実行できているかどうかチェックしてみてください。
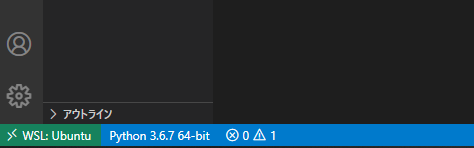
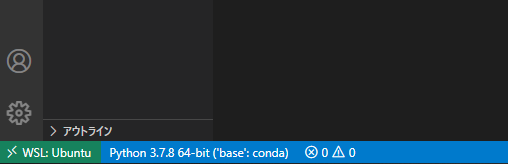
もしデフォルトのPythonが選択されている場合は、左下の「Python 3.〇.〇 64-bit」の部分をクリックしてPythonインタープリターの選択画面を表示させて、conda環境のPythonを選択してください。
Jupyter Notebook の実行
次に、Jupyter Notebookを実行してみましょう。
先ほどのPythonのプログラムではPythonのスクリプトファイル(.py)にコードを書いてそれを実行するというスタイルでしたが、実際のデータ解析においては「一つ一つ処理を実行していって、出てくる結果を見ながら進めていく」ということが多いと思います。(もちろん、すでに完璧なパイプラインがあるのなら、Pythonスクリプトを実行するだけで済むのでしょうが…)
その時に役に立つのがJupyter Notebookです。Jupyter Notebookを使えば、一つ処理を行うごとに結果を確認して、次の処理に進んでいくことができます。さらに、解析のステップごとに処理をやり直したり、Pythonのスクリプトとその結果を同じファイルに保存してほかの人と解析結果を共有したりと様々な利点があります。おそらくデータサイエンスの世界ではデファクトスタンダードでしょう。そのJupyter Notebookをバイオインフォマティクスの領域でも使ってみます。
Jupyter Notebook自体はAnacondaに含まれており、Visual Studio CodeのPython拡張機能をインストールすることでVisual Studio Codeから使うこともできます。Minicondaをインストールした場合は、Jupyter Notebookに必要なモジュールがインストールされていないので、途中でインストールを促されると思います。その時は、指示に従ってcondaからインストールしてください。
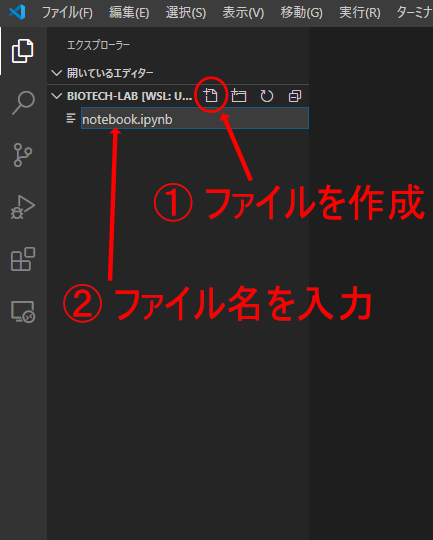
まず、Pythonスクリプトファイルの時と同様に、左側のエクスプローラにJupyter Notebookのファイル(.ipynb)を作成します。この拡張子により自動的にJupyter Notebookと認識してくれるので、これを開けば自動的にJupyter Notebookが開きます。
Jupyter Notebookでは、セルと呼ばれるブロックごとにプログラムを書いて実行していきます。下のようにプログラムを書いて実行してみましょう。
この時、Pythonインタープリターの選択画面が出たら、conda環境のPythonを選択してください。
ちなみに、Jupyter Notebookはここで示したように先にNotebookファイルを作成せずに直接起動することもできますが、Notebookファイルを作成していないと相対パスを用いて外部ファイルを指定することができないので、ここでは先にNotebookファイルを作成しています。
まとめ
以上のような方法で、同一のWindowsアプリケーションの中で、Linuxの操作も完結することができ、さらにPythonのスクリプトも書くことができます。さらに、作業ディレクトリはWindows内とすることもでき、実質的にはWindows内でゲノム解析をしているような状況にすることができると思います。また、データ解析では標準的なJupyter Notebookも用いることができます。
それでは、今後はこの環境を用いてゲノム解析を進めていこうと思います。









コメント