Pydicomを用いてDICOMファイルを開き、画像とメタデータを取得する方法を説明します。
[toc]DICOMファイルを読み込む
ここではテストデータの「CT_small.dcm」を用います。get_testdata_file関数でファイルパスを取得し、dcmread関数でファイルをFileDatasetオブジェクトとして読み込みます。
import pydicom
path = pydicom.data.get_testdata_file('CT_small.dcm')
dataset = pydicom.filereader.dcmread(path)
print(type(dataset))
<class 'pydicom.dataset.FileDataset'>
画像を取得する
Datasetクラス(FileDatasetクラスの継承元クラス)のpixel_arrayプロパティを用いることで画像データがNumPyのndarrayとして取得できます。画像データがndarrayとして得られれば、OpenCVでもmatplotlibでもPillowでも好きなものを使って表示することができます。
ここでは、matplotlibを用いて画像を表示する例を示します。
import pydicom
path = pydicom.data.get_testdata_file('CT_small.dcm')
dataset = pydicom.filereader.dcmread(path)
img = dataset.pixel_array
import matplotlib.pyplot as plt
fig, ax = plt.subplots()
ax.imshow(img, cmap='gray')
ax.set_axis_off()
plt.show()
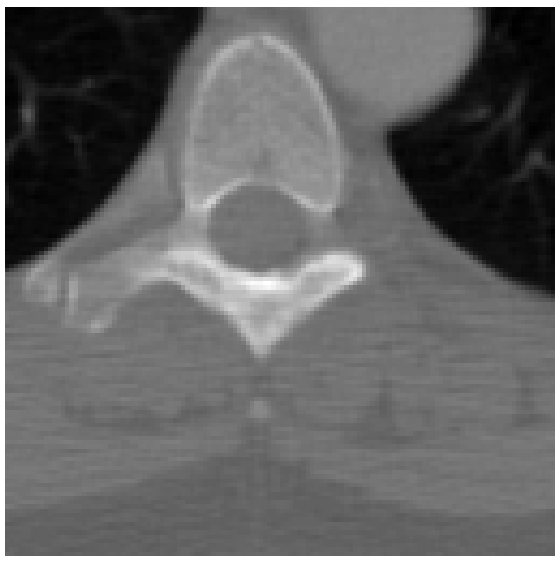
なお、pixel_arrayプロパティで得られる画像データはカラーの場合はRGB画像となりますので、matplotlibではそのまま表示できますが、OpenCVを用いる場合は気を付けてください。
import pydicom
path = pydicom.data.get_testdata_file('US1_UNCI.dcm')
dataset = pydicom.filereader.dcmread(path)
img = dataset.pixel_array
import matplotlib.pyplot as plt
fig, ax = plt.subplots()
ax.imshow(img)
ax.set_axis_off()
plt.show()
メタデータを取得する
メタデータの取得方法
DICOMファイルには画像データとそれに付随する患者情報・モダリティ・撮影装置名などのメタデータが含まれています。このメタデータにアクセスする方法を説明します。
PydicomではDICOMデータをFileDatasetオブジェクトとして読み込みますが、このFileDatasetクラスはPythonの辞書型を継承したクラスであり、画像データやメタデータは辞書のような形で格納されています。DICOM規格では画像データやメタデータのtag、name、keywordが定められていて(Registry of DICOM Data Elements)、そのtagをDatasetクラス(FileDatasetクラスの継承元クラス)の辞書のkeyとして指定することで、そのデータをDataElementクラスのオブジェクトとして取得することができます。
例えば、患者名を表すデータをRegistry of DICOM Data Elementsから探すと、tagが(0010,0010)、keywordがPatientNameであることが分かります。なお、DICOMのtagは16進数で表されているので、(0010,0010)で表されるtagは、(0x0010, 0x0010)として、16進数であることを表す’0x’を追加する必要があります。
import pydicom
path = pydicom.data.get_testdata_file('CT_small.dcm')
dataset = pydicom.filereader.dcmread(path)
data = dataset[(0x0010, 0x0010)]
print(type(data))
<class 'pydicom.dataelem.DataElement'>
これで患者名を表すデータがDataElementオブジェクトとして得られました。ここから実際の患者名を取得するには、DataElementクラスのvalueプロパティを用います。
import pydicom
path = pydicom.data.get_testdata_file('CT_small.dcm')
dataset = pydicom.filereader.dcmread(path)
data = dataset[(0x0010, 0x0010)]
print(data.value)
CompressedSamples^CT1
あくまでもサンプルデータを用いているので患者名らしくない名前になっていますが、これで患者名が取得できました。
しかし、Datasetオブジェクトの辞書のkeyが(0x0010, 0x0010)のような数字の羅列では後からコードを見直した時や、第三者がコードを読んだときに非常に読みにくくなってしまいますよね?これでは、何の値を取得しているのかがわからずに後からコードのメンテナンスが困難になったり、バグの温床になったりしてしまいます。それを解消するためにDatasetクラスは取得したいデータのkeywordを属性として指定することができるようになっています。先ほどの患者名を表すデータのkeywordはPatientNameだったので、以下のようになります。
import pydicom
path = pydicom.data.get_testdata_file('CT_small.dcm')
dataset = pydicom.filereader.dcmread(path)
name = dataset.PatientName
print(name)
CompressedSamples^CT1
この方法では、DataElementオブジェクトのvalueが直接取得できます。これでコードも非常に読みやすくなりましたね。
DataElementオブジェクトのvalueプロパティについて
上の例では、患者名がDataElementオブジェクトのvalueプロパティに含まれるのですが、valueプロパティで取得されるものはそれぞれのデータの内容に応じたデータ型のオブジェクトとなっています。例えば、患者名であればPersonName3オブジェクトとなります。
文字列オブジェクトとして患者名を取得するには、DataElementオブジェクトのrepvalプロパティを用います。もちろん、標準出力に出すだけなら上の例のようにvalueをprint関数の引数に渡すだけで適切に処理してくれます。
DICOMファイルのメタデータ一覧を取得する
DICOM規格で定義されているtagやkeywordの一覧はRegistry of DICOM Data Elementsにありますが、実際のDICOMファイルはこのすべてを持っているわけではありません。例えば、CT画像を表すDICOMデータであれば、MRIに関するメタデータは持っていません。読み込んだDICOMファイルが持っているkeywordの一覧は、Datasetクラスのdirメソッドで取得することができます。
import pydicom
path = pydicom.data.get_testdata_file('CT_small.dcm')
dataset = pydicom.filereader.dcmread(path)
print(dataset.dir())
['AccessionNumber', 'AcquisitionDate', 'AcquisitionNumber', 'AcquisitionTime', 'AdditionalPatientHistory', 'BitsAllocated', 'BitsStored', 'Columns', 'ContentDate', 'ContentTime', 'ContrastBolusAgent', 'ContrastBolusRoute', 'ConvolutionKernel', 'DataCollectionDiameter', 'DataSetTrailingPadding', 'DistanceSourceToDetector', 'DistanceSourceToPatient', 'Exposure', 'ExposureTime', 'FilterType', 'FocalSpots', 'FrameOfReferenceUID', 'GantryDetectorTilt', 'HighBit', 'ImageComments', 'ImageOrientationPatient', 'ImagePositionPatient', 'ImageType', 'InstanceCreationDate', 'InstanceCreationTime', 'InstanceCreatorUID', 'InstanceNumber', 'InstitutionName', 'KVP', 'Laterality', 'Manufacturer', 'ManufacturerModelName', 'Modality', 'OtherPatientIDsSequence', 'PatientAge', 'PatientBirthDate', 'PatientID', 'PatientName', 'PatientPosition', 'PatientSex', 'PatientWeight', 'PhotometricInterpretation', 'PixelData', 'PixelPaddingValue', 'PixelRepresentation', 'PixelSpacing', 'PositionReferenceIndicator', 'ReconstructionDiameter', 'ReferringPhysicianName', 'RescaleIntercept', 'RescaleSlope', 'Rows', 'SOPClassUID', 'SOPInstanceUID', 'SamplesPerPixel', 'ScanOptions', 'SeriesDate', 'SeriesInstanceUID', 'SeriesNumber', 'SeriesTime', 'SliceLocation', 'SliceThickness', 'SoftwareVersions', 'SpacingBetweenSlices', 'SpecificCharacterSet', 'StationName', 'StudyDate', 'StudyDescription', 'StudyID', 'StudyInstanceUID', 'StudyTime', 'TableHeight', 'TimezoneOffsetFromUTC', 'XRayTubeCurrent']
また、dirメソッドは引数に文字列を指定することで、その文字列を含むものを絞り込んで検索することもできます。例えば、患者情報に関するメタデータとして何が含まれているかを調べるには、dirメソッドの引数に’patient’を指定することで次のようにできます。
import pydicom
path = pydicom.data.get_testdata_file('CT_small.dcm')
dataset = pydicom.filereader.dcmread(path)
print(dataset.dir('patient'))
['AdditionalPatientHistory', 'DistanceSourceToPatient', 'ImageOrientationPatient', 'ImagePositionPatient', 'OtherPatientIDsSequence', 'PatientAge', 'PatientBirthDate', 'PatientID', 'PatientName', 'PatientPosition', 'PatientSex', 'PatientWeight']
これで、’patient’という文字列が含まれたメタデータのkeyword一覧が取得できました。
DICOMファイルのデータ構造
DICOMファイルにおける画像データの扱い
実は、DICOMファイルでは画像データもメタデータも区別なく格納されています。先ほど、メタデータにはtagやkeywordが割り当てられていて、そこからデータを取得できると説明しましたが、画像データもまったく同様にtagやkeywordが割り当てられていて、メタデータと同等の扱いになっています。
画像データのtagは(7FE0,0010)で、keywordはPixelDataなので、次のようにして画像データの本体を取得できます。
import pydicom
path = pydicom.data.get_testdata_file('CT_small.dcm')
dataset = pydicom.filereader.dcmread(path)
img = dataset.PixelData
print(type(img))
<class 'bytes'>
一番最初に説明したpixel_arrayプロパティは、getterでこの値をnumpy.ndarrayに変換して返していたくれたのです。ただし、pixel_arrayプロパティにはsetterが定義されていないので、画像データを編集してDICOM形式で保存する際は直接このPixelDataを操作する必要が出てきます。
DICOMファイルのデータ構造
ここまで理解していればDICOMファイルを扱ううえで困ることはないのですが、最後にDICOMファイルのPydicomにおけるデータ構造について技術的な面から説明しておきます。
- まずDICOMファイルをFileDatasetオブジェクトとして読み込みます。DICOMファイルには以下のような情報が含まれています。
- DICOMファイルに関する情報(ファイル名など)
- データ本体:keyをBaseTagオブジェクト、valueをDataElementオブジェクトとする辞書構造
- データ本体のDataElementオブジェクトにはkeyword, name, VR, VM, valueなどの要素が含まれています。
- そのうち、データ自体を表すvalueにはpydicom.valuerepモジュールのオブジェクトかSequenceオブジェクトのいずれかでデータが格納されています。
- Sequenceオブジェクトには、Datasetオブジェクトがリスト形式で格納されています。
- 一つ一つのDatasetオブジェクトには、先ほどと同様にkeyをBaseTagオブジェクト、valueをDataElementオブジェクトとする辞書構造でデータが格納されています。
- データ本体のDataElementオブジェクトには… (以下、繰り返し)
上図のように、DICOMファイルは入れ子構造でデータを格納していることが分かります。








コメント