「○○であるかどうか」を予測するモデルとしてロジスティック回帰モデルがあります。実際には○○である確率を求めるものですが、それをもとに○○かどうかを判定することができるので、これは回帰分析でありながら「○○かどうか」の2つの状態の分類を表すモデルといえます。ここではscikit-learnでサンプルデータとして用意されている乳房腫瘍の細胞診で得られた結果から「悪性 / 良性」の判定を、ロジスティック回帰モデルを用いて行ってみましょう。
開発環境
- Python 3.7.11
- scikit-learn 0.24.2

ロジスティック回帰モデルとは
ロジスティック回帰モデルは目的変数が「生存 / 死亡」「陽性 / 陰性」「悪性 / 良性」のような2値の質的変数しかとらない場合、つまり「○○であるかどうか」を表す場合の線形回帰モデルに相当します。「生存 / 死亡」のようなケースは医学研究においてよく出てくるので、ロジスティック回帰モデルは医学研究で頻繁に目にするモデルになります。「○○であるかどうか」を表すロジスティック回帰モデルで出力される結果は「○○である確率」ですが、例えばその確率を閾値(0.5など)によって区切ることで「○○であるかどうか」を判定することができるので、回帰モデルでありながら分類モデルのように扱うこともできます。
なお、説明変数は量的変数でも質的変数でもどちらでも構いません。また今回は扱いませんが、目的変数が3つ以上の場合は多項ロジットモデルとなります。
詳細は以下の記事もご覧ください。
データセットの取得と確認
ここではロジスティック回帰モデルを用いた解析の例として、乳房腫瘍の細胞診で得られたデータと「悪性 / 良性」の診断結果から成るデータセットを用います。このデータセットはscikit-learnのサンプルデータとして提供されておりload_breast_cancer関数で取得できます。まずはこれから解析するデータを俯瞰してみましょう。なおここで取得するデータ形式はBunchオブジェクトですが、Bunchオブジェクトについては次の記事をご覧ください。
データ解析を行う際の基本として、まずはそのデータの全体像を見てみましょう。データ全体を見るためにはDataFrameとして取得した方が操作しやすいので、load_breast_cancer関数でas_frame=Trueに設定して取得します。
# データの取得・確認
from sklearn.datasets import load_breast_cancer
breast_data = load_breast_cancer(as_frame=True)
print(breast_data.frame.describe())
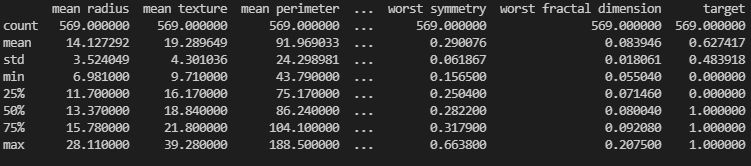
frameキーの値に説明変数と目的変数の両方が統合されたDataFrameが格納されているので、そのdescribeメソッドで基本的な統計量をまとめて表示させてデータを要約しています。countはすべて569であることから欠損値はなさそうです。一方で各項目のmeanとstdの値はバラバラであることから標準化した方がいいかもしれません。
なお、それぞれの項目の意味は次のようになります。
- mean radius : 細胞核の半径の平均
- mean texture : 細胞核のテクスチャの平均(テクスチャ:グレースケールの画素値の標準偏差)
- mean perimeter : 細胞核の外周長の平均
- mean area : 細胞核の面積の平均
- mean smoothness : 細胞核のなめらかさの平均
- mean compactness : 細胞核のコンパクトさの平均
- mean concavity : 細胞核の輪郭の凹凸の程度の平均
- mean concave points : 細胞核の輪郭の凹凸の個数の平均
- mean symmetry : 細胞核の対称性の平均
- mean fractal dimension : 細胞核のフラクタル次元の平均
- radius error : 細胞核の半径の誤差
- texture error : 細胞核のテクスチャの誤差
- perimeter error : 細胞核の外周長の誤差
- area error : 細胞核の面積の誤差
- smoothness error : 細胞核のなめらかさの誤差
- compactness error : 細胞核のコンパクトさの誤差
- concavity error : 細胞核の輪郭の凹凸の程度の誤差
- concave points error : 細胞核の輪郭の凹凸の個数の誤差
- symmetry error : 細胞核の対称性の誤差
- fractal dimension error : 細胞核のフラクタル次元の誤差
- worst radius : 細胞核の半径の最悪値
- worst texture : 細胞核のテクスチャの最悪値
- worst perimeter : 細胞核の外周長の最悪値
- worst area : 細胞核の面積の最悪値
- worst smoothness : 細胞核のなめらかさの最悪値
- worst compactness : 細胞核のコンパクトさの最悪値
- worst concavity : 細胞核の輪郭の凹凸の程度の最悪値
- worst concave points : 細胞核の輪郭の凹凸の個数の最悪値
- worst symmetry : 細胞核の対称性の最悪値
- worst fractal dimension : 細胞核のフラクタル次元の最悪値
- target : 悪性もしくは良性 (0:悪性、1:良性)
データの前処理
データを取得したらまずは解析に適した状態にするための前処理が必要になります。先ほど見たようにここでは説明変数の平均・標準偏差がそろっていないので、それをそろえるために標準化を行っておきましょう。ここではscale関数を用いて標準化を行います。
# データの標準化
from sklearn.preprocessing import scale
standardized_breast_data = scale(breast_data.data)
データセットを訓練用データと評価用データに分ける
機械学習で作成したモデルを評価するためには、モデル作成(訓練)に用いたデータセットとは別のデータセットで評価する必要があります。そうでないと、訓練に用いたデータセットでしか成り立たないモデルなのか、一般的な状況でも成り立つモデルなのかが分からないからです。scikit-learnではデータセットを指定した割合で訓練用と評価用のデータにランダムに分ける関数としてtrain_test_split関数が用意されているので、ここではこの関数を用いて20%を評価用のデータに指定して分割してみましょう。なお、説明変数には先ほど標準化したデータを指定します。
# 訓練用データと評価用データに分割
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(standardized_breast_data, breast_data.target, test_size=0.2, random_state=100)
これで訓練用データ(X_train, y_train)と評価用データ(X_test, y_test)に分割できました。ただし、X_***が説明変数でy_***が目的変数です。なお、ここでは乱数のシードを100に固定しています。この値は任意でいいのですが、機械学習のモデルの性能を比較する場合は乱数をデータセットの分割方法をそろえる必要があるので気をつけましょう。
ロジスティクス回帰モデルで予測を行ってみる
ここまで準備できたら、早速ロジスティック回帰モデルを作成してみましょう。まずモデル(LogisticRegressionクラス)をインスタンス化して、そのfitメソッドに訓練用のデータを指定することでそのデータに合うモデルが生成されます。
# モデル作成
from sklearn.linear_model import LogisticRegression
model = LogisticRegression()
model.fit(X_train, y_train)
生成されたモデルの評価を行う
訓練データでモデルを作成したら、その評価を行いましょう。モデルの評価方法には様々な方法がありますが、分類モデルの評価には正しく分類できている割合を表す正解率が有効な指標となります。LogisticRegressionクラスのscoreメソッドで正解率を求めてみましょう。
# モデルの評価
print('訓練データ:' + str(model.score(X_train, y_train)))
print('評価データ:' + str(model.score(X_test, y_test)))
訓練データ:0.9934065934065934 評価データ:0.9736842105263158
これにより訓練データから得られた結果は正解率0.99という高い値を示し、評価用データでは0.97と若干正解率が下がっていますがこちらも十分良好な結果と言えそうです。
なお、predict_probaメソッドに説明変数を指定すればその腫瘍が良性である確率が算出され、predictメソッドではそれをもとに良性・悪性を予測することができます。
コードまとめ
最後に上記のコードをまとめてみましょう。
from sklearn.datasets import load_breast_cancer
from sklearn.preprocessing import scale
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression
# データの取得・確認
breast_data = load_breast_cancer(as_frame=True)
print(breast_data.frame.describe())
# データの標準化
standardized_breast_data = scale(breast_data.data)
# 訓練用データと評価用データに分割
X_train, X_test, y_train, y_test = train_test_split(standardized_breast_data, breast_data.target, test_size=0.2, random_state=100)
# モデル作成
model = LogisticRegression()
model.fit(X_train, y_train)
# モデルの評価
print('訓練データ:' + str(model.score(X_train, y_train)))
print('評価データ:' + str(model.score(X_test, y_test)))
補足:ロジスティック回帰分析で各因子の有意性を求めるには?
上記ではロジスティック回帰モデルを採用して、検査データから悪性/良性の結果を予測するモデルを作成しました。しかし、実際には予測モデルを作成することが目的ではなく、各因子のうち何が有意に結果に関わっているのかを調べる目的でロジスティック回帰分析を行うこともありますよね?むしろ医学研究におけるロジスティック回帰分析と言えば、各因子の有意差の有無を統計的に調べることを言うといっても過言ではないかもしれません。では、上記の予測モデルをそのような目的で使うことはできるでしょうか?
scikit-learnで作成したロジスティック回帰モデルの係数と切片は次のようにして取得可能です。
print(model.coef_) # 係数
print(model.intercept_) # 切片
ただし、scikit-learnではそれぞれの係数のp値を取得することはできません。scikit-learnはあくまでも機械学習で与えられたデータから結果を予測することを目的としたライブラリなので、各因子の関係を示すことは目的としていないのです。そのため、より正確に予測できるモデルが作れればよいのであって、scikit-learn的にはその係数のp値は何であってもあまり重要ではないのです。
ロジスティック回帰分析で各因子のp値を求めて統計的検定を行うためには、統計モデルのライブラリであるstatsmodelsを使う必要があります。詳細は以下の記事をご覧ください。






コメント